2015-12-01 16:03:08
看到新版PyMOL的体数据渲染功能增强了, 就想试一试.
最新版的PyMOL在Windows下目前还没有直接的安装文件供下载, 需要自己安装. 参考下面的资料
将安装方法简记如下:
-
下载安装最新版的Python 2.x.
安装时Advanced选项下选择编译为.pyc, 并添加路径到环境变量 -
下载安装PyMOL需要的包Pmw和Numpy+MKL
由于新版本的Python已经自带了pip, 所以无须再单独下载安装pip了. 直接使用pip安装上面两个包即可. -
下载安装PyMOL
直接使用pip安装 -
下载安装PyMOL_Launcher. 这个包不是必须的, 安装不安装都可以.
-
下载安装各种脚本. 这也不是必须的, 如果需要就安装.
下载时注意选择和自己的系统一致的文件. 安装完成后, 双击/Python主目录/Scripts/pymol.cmd即可启动pyMOL.
根据我的测试, 在Windows下, PyMOl 1.7.x系列中支持体数据渲染功能的最高版本为1.7.0, 但功能有限. PyMOL 1.8.x系列确实支持体数据渲染, 可惜的是, 上面网站编译的PyMOL没有包含VMD的Molfile插件, 因此无法打开GAUSSIAN .cube格式的密度文件, 不方便使用. 当然可以自己重新编译PyMOL, 使其包含VMD的Molfile插件, 但这实在不是一件容易的事.
如果你确实需要使用PyMOL的体数据渲染功能, 并且一定要使用1.8.x系列的版本, 一个解决办法是不使用.cube格式的密度文件, 而是使用PyMOL原生支持的密度文件格式. 由于所用处理程序的不同, 密度文件的格式也很繁多, 大致分为二进制格式和文本格式. 二进制格式的优点是存储空间小, 读取速度快, 但不容易生成, 且在不同系统上存在兼容性问题. 目前主要使用的二进制格式是.ccp4以及.omap. 相比.ccp4, .omap格式的文件可能最小. 文本格式的类型不多, 主要有前面提到的.cube格式, .ezd格式, .xplor(或称.cns)格式. 对未包含VMD Molfile插件的PyMOL 1.8.x, 无法读入.cube和.ezd格式的密度文件, 但可读入.xplor格式的文件, 这样的话我们可以先将.cube格式转换为.xplor格式, 就可以利用PyMOL进行渲染了. 虽然麻烦点, 但总算能达到目的. 这些格式的说明可参考
那如何使用体数据渲染呢? 可参考
保存图像时需要使用set ray_volume, on, 否则不会保存体数据的显示. 另一种解决方法可参考Volume - ray - rendering.
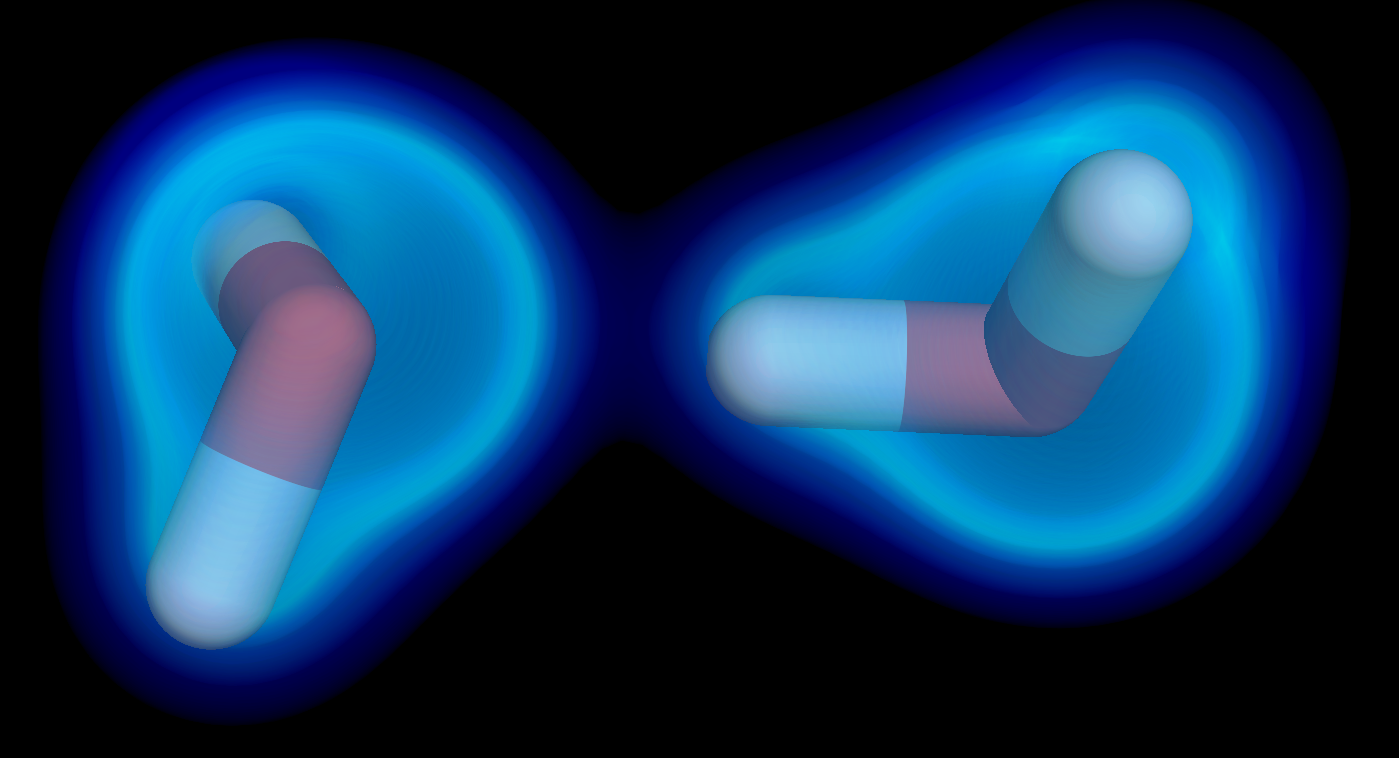
下面是水分子二聚体的电子密度图
此外, 另一款支持体数据渲染的软件是Chimera, 但果可能稍差. PovRay, ParaView或yt也有办法实现体渲染, 具体方法可参考
