- 2019-02-12 17:32:07
英文的cluster这个词, 中文可以翻译成团簇, 也可以翻译成聚类. 虽然数学上来说实质相同, 当具体使用的时候还是有些不同的, 适当加以区分可以更明确, 避免一些模糊之处.
在MD的语境下, 我们说进行团簇分析, 一般指的是, 给定一个构型, 其中包含多个原子, 我们按一定的规则(常用的是距离)将这些原子归属到不同的聚集体, 也就是团簇中, 这样可以将体系划分为一些团簇, 对每个团簇进行分析, 获取一些信息. 这种做法和我们根据一堆原子相互间的成键, 将它划分为一个一个的分子是一样的. 在这种情况下, 我们的规则就是原子间是否成键, 团簇就是分子. 如果两个原子间成键, 它们就属于同一分子, 也就归属到同一团簇.
可以将上面团簇分析的概念推广一下. 每个原子可以代表抽象的对象, 而不一定是原子或粒子, 可以是任何你定义的东西, 如分子, 蛋白, 体系, 数字, 字符串, 图片, 个人, 国家, 星球. 只要你能在这些对象之间定义一个判断归属的量(可称为广义的距离, 常用的如马氏距离, 编辑距离等, 也可以是任何其他属性, 蛋白不同构象之间的rmsd, 图片之间的相似度, 人与人之间是否联系等), 按照一定的方法, 我们就可以将这些对象归属到不同的类别中去, 这就是聚类分析. 得到的聚类是一些具有类似属性的对象.
物以类聚, 人以族分. 各类物种划分, 皆是如此.
GROMACS中涉及到聚类分析的程序有两个. gmx clustize用于狭义的团簇分析, 也就是前面所说的将一堆原子划分为不同的团簇; gmx cluster可进行广义的聚类分析, 但程序实现的功能只是根据RMSD对蛋白的不同构象进行聚类分析, 从而将大量蛋白构象划分为不同的类别. 这种作法一般用于多肽的折叠研究, 对得到的轨迹进行分析, 看多肽主要存在哪些构象类型.
假定我们对蛋白进行了一段时间的模拟, 得到了轨迹. 如果是个大的蛋白, 除loop区外, 构象一般不会有太大变化, 所以我们也不大关心其聚类分析. 但如果是个小的多肽链, 那么它的构象在整个模拟过程中可能会发生很多变化, 二级结构也可能变来变去. 这种情况下我们就可以对这个多肽链的所有构象进行聚类分析, 看看它到底有那些比较稳定的构象, 各个构象的分布如何. 这就是使用gmx cluster的目的.
所以对于gmx cluster进行的聚类分析来说, 对象就是不同构象的蛋白, 所有对象的集合就是整条轨迹中所有的蛋白构象, 归属标准(距离)就是RMSD, 得到的每个聚类就是一些RMSD互相接近的蛋白构象, 对同一聚类的构象进行叠合所得的平均构象可以作为这个聚类的代表.
为此, 我们需要每两个构象之间的RMSD矩阵, 基于它才能进行后面的聚类分析.
gmx cluster有两个输出选项, 默认情况下RMSD矩阵的输出为rmsd.xpm文件. 这是xpm格式的数据, 方便直接查看, 但不方便用于其他作图软件. 我们可以将其转换为常用的数据文件, 但这样得到的数据精度有限. gmx cluster还有一个输出选项-bin, 可以直接将RMSD矩阵输出到rmsd.dat文件. 但这却是个二进制格式的文件, 没法直接用文本查看. 所以我们需要将其转换为普通的文本数据. 方法当然很多了, 任何支持二进制文件的编程语言都可以. c, fortran, python, matlab, 这些常用的语言都可以. 你喜欢哪个就用那个, 你熟悉哪个就用那个.
可如果你非要嫌那些语言过于笨重, 是杀鸡的牛刀, 打蚊子的高射炮, 那我们可以用shell自带的工具. 其中一个就是od, 它可以将任意文件以任意格式输出, 二进制的当然不在话下. 所以如果我们得到的是单精度的rmsd.dat, 使用
od -f -v rmsd.dat就可以将其转换为普通数据格式.
当然这样得到的数据还没有排列好, 我们需要一点格外的代码将数据排列出我们需要的格式, 可以是x, y, z格式的, 也可以是矩阵格式的.
有了数据就好办了, 你可以使用自己喜欢的软件来作图, 还可以选择使用你喜欢的颜色映射方案.
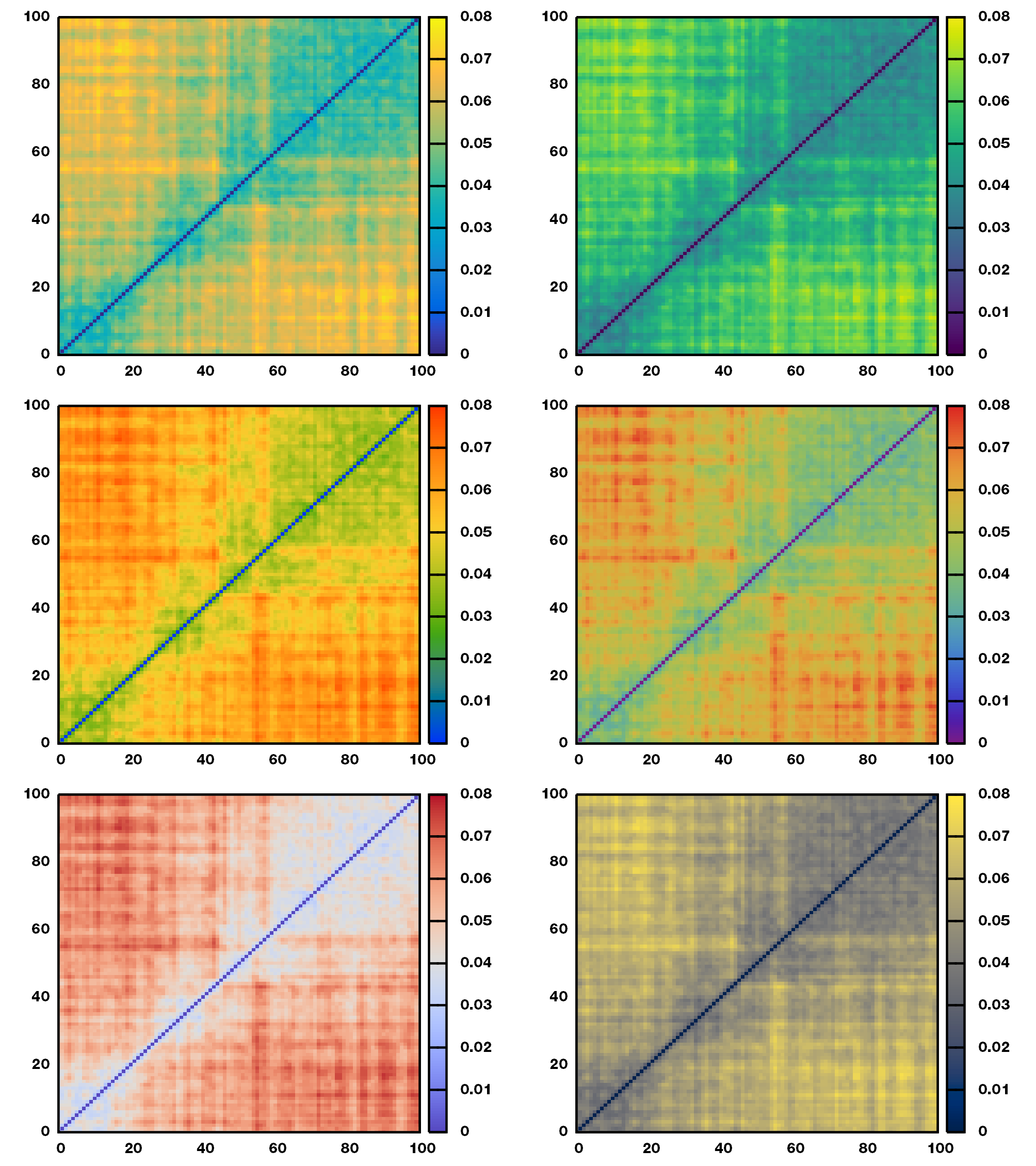
美美哒.
什么, 不够美? 那试试三维效果, 或者使用地图的晕染效果, 或者使用更真实的光照渲染.
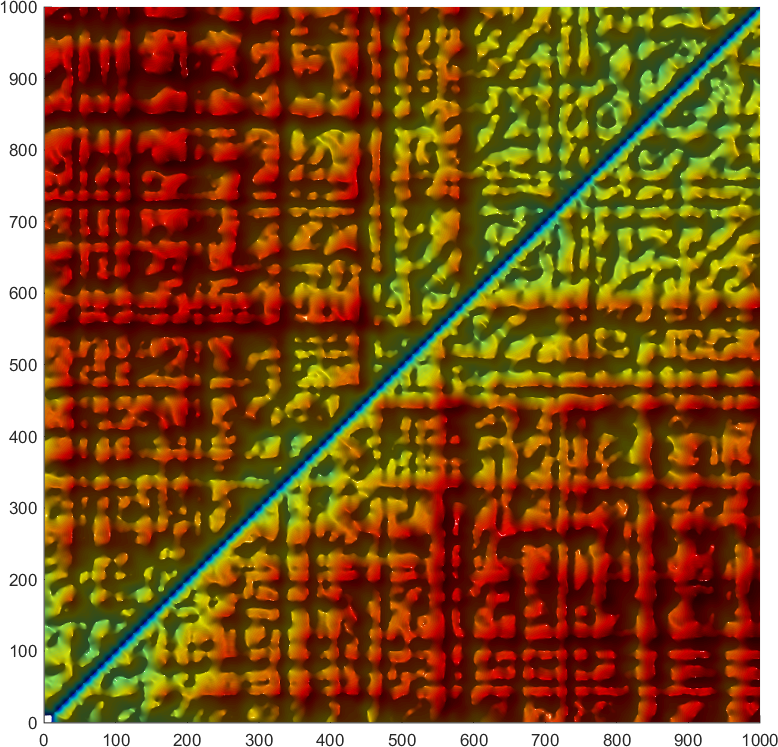
怎么, 不完美? 那我们加上蛋白构象. 要颜值有颜值, 要直观有直观.
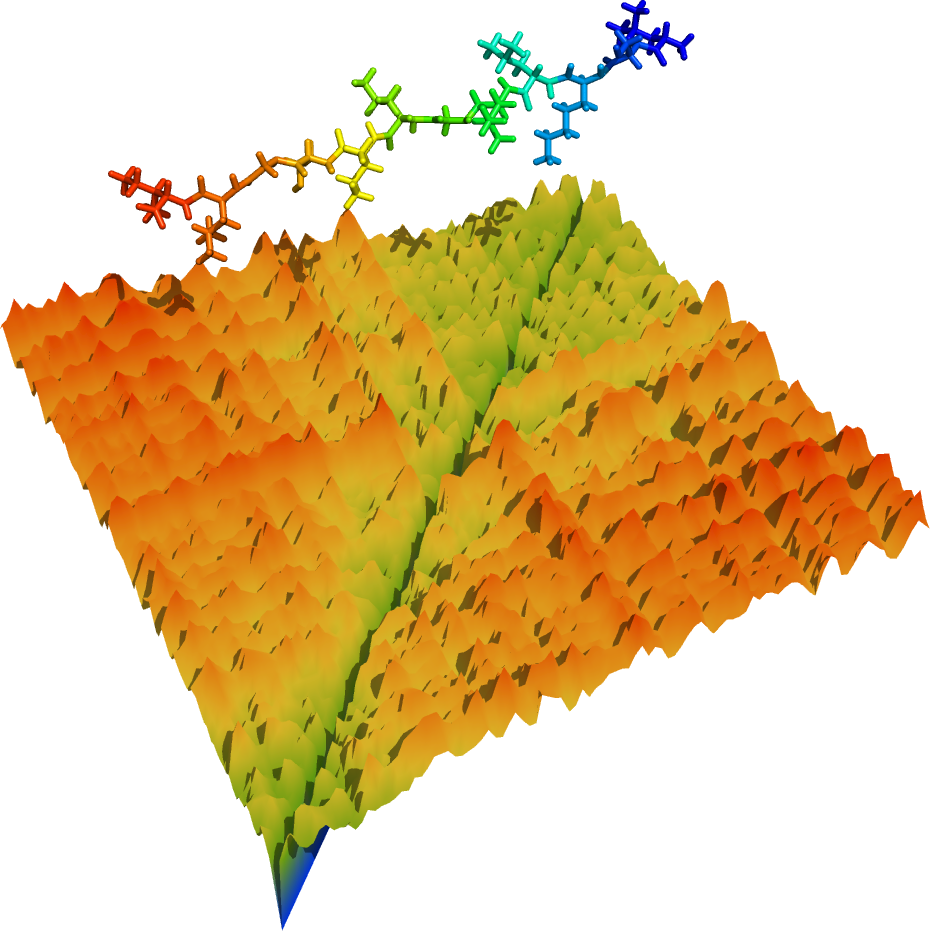
peeeeeerfect.
这不, 连自己都感动了.
等千帆阅尽, 万图览毕, 返璞归真, 你才发现, 其实这些也便是如此. 黑白灰, 就像老照片, 也很耐看, 甚至更耐人寻味.
此图暂缺
主题呢? 主题呢?
是啊, 我们的主题呢? 不是要做聚类分析的么? 怎么走着走着就着了化妆的道, 入了美颜的魔, 忘了初心? 那还不赶快回来. 好吧, 你也不得不承认, 有些事情就是比另一些事情更吸引人, 更容易让人沉迷, 我们对此要有戒心啊. 有戒才能定, 有定才生慧. 观自在菩萨早就教导过我们, 色不异空, 空不异色, 色即是空, 空即是色, 空中无色, 色中无空, 五蕴皆空, 方能度一切苦厄, 得无上正道, 究竟涅槃.
gmx cluster中提供了几个聚类分析的算法, 但实际上聚类算法非常多, 因为聚类分析是机器学习中最基础的东西, 所有涉及机器学习的领域都会用到. 这几年机器学习, 人工智能大热, 聚类分析的算法也是层出不穷. sklearn中就有很多的示例.
聚类分析的方法太多了, 选的心累, 我们就此打住, 只用最简单最基础的层次聚类, 用matlab很容易实现, 结果却需要好好理解一下.
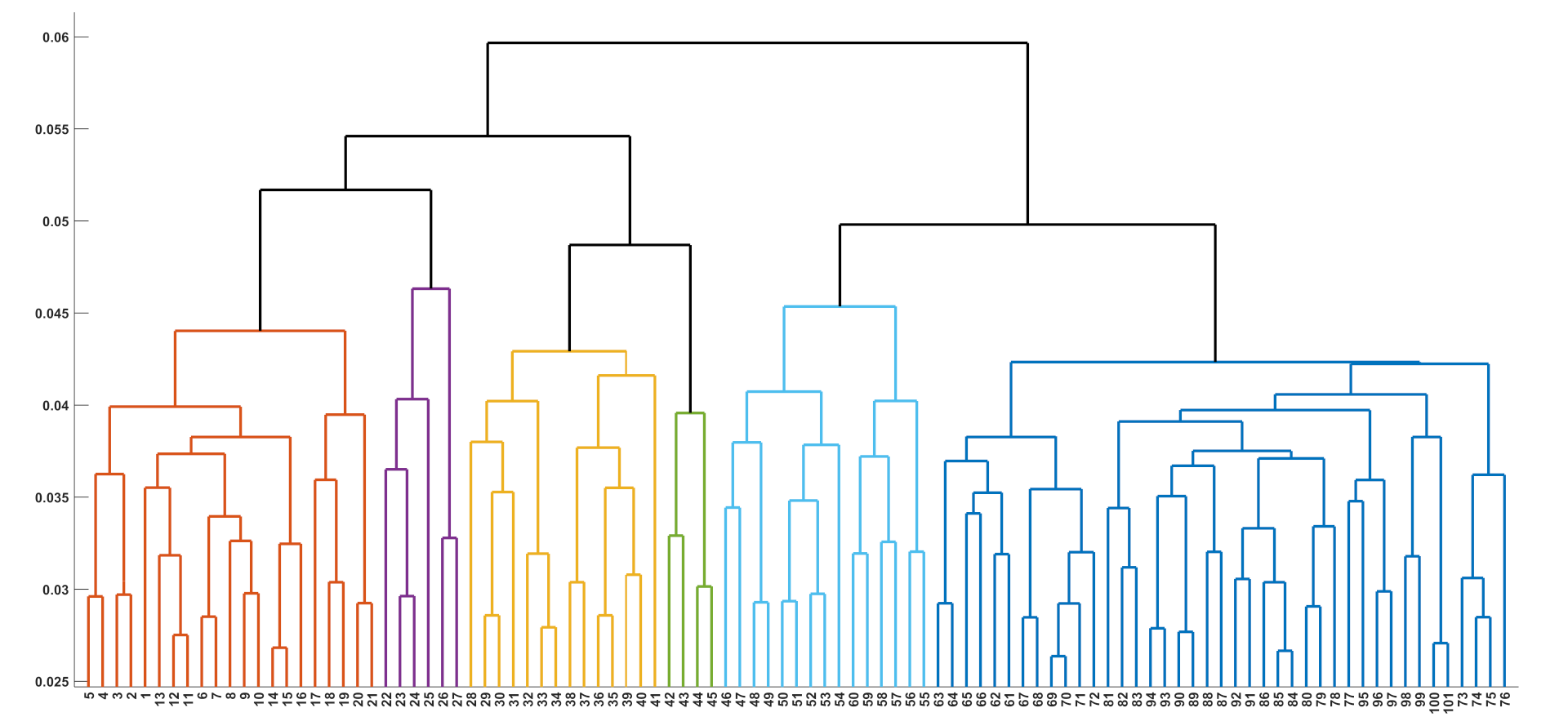
有了这个图, 你想划分为几个聚类就划分为几个聚类, 每个聚类中的对象, 也标识得清清楚楚.
到这里, 我们的主题基本算是完成了. 那就 以上 吧.
以上.
