- 2021-04-21 22:24:44
对于蛋白模拟轨迹, 可以方便地使用gmx rama计算肽键的二面角φ和ψ, 用于绘制Ramachandran图, 也就是常说的拉氏图. 这种图常用于表征蛋白的二级结构, 有时也用于评估蛋白结构是否合理.
gmx rama输出的rama.xvg文件包含的内容如下:
@ title "Ramachandran Plot"
@ xaxis label "Phi"
@ yaxis label "Psi"
@TYPE xy
@ with g0
@ s0 linestyle 0
@ s0 color 1
@ view 0.2, 0.2, 0.8, 0.8
@ world xmin -180
@ world ymin -180
@ world xmax 180
@ world ymax 180
@ xaxis tick on
@ xaxis tick major 60
@ xaxis tick minor 30
@ yaxis tick on
@ yaxis tick major 60
@ yaxis tick minor 30
@ s0 symbol 2
@ s0 symbol size 0.4
@ s0 symbol fill 1
-131.224 148.225 THR-2
-150.907 147.872 CYS-3
-138.757 146.255 CYS-4
-69.0707 -23.5039 PRO-5
-143.503 169.489 SER-6
(略)
-86.3428 156.502 PRO-41
-58.009 -4.88865 GLY-42
-104.401 -23.6048 ASP-43
-121.252 58.847 TYR-44
-96.68 8.08769 ALA-45
-122.848 142.18 THR-2
-142.218 148.986 CYS-3
-136.066 149.023 CYS-4
-76.9576 -13.3019 PRO-5
-151.13 169.091 SER-6
(略)
-87.8832 174.68 PRO-41
-82.3377 -6.16846 GLY-42
-99.6951 -8.30811 ASP-43
-127.731 35.0559 TYR-44
-80.2416 10.7315 ALA-45
(略)很容易看出来, 文件中给出了每帧轨迹中每个残基的(φ,ψ), 但不包括最后一个残基, 因为这两个二面角是两个相邻残基之间的肽键对应的二面角.
最简单的绘制方法就是直接绘制所有点
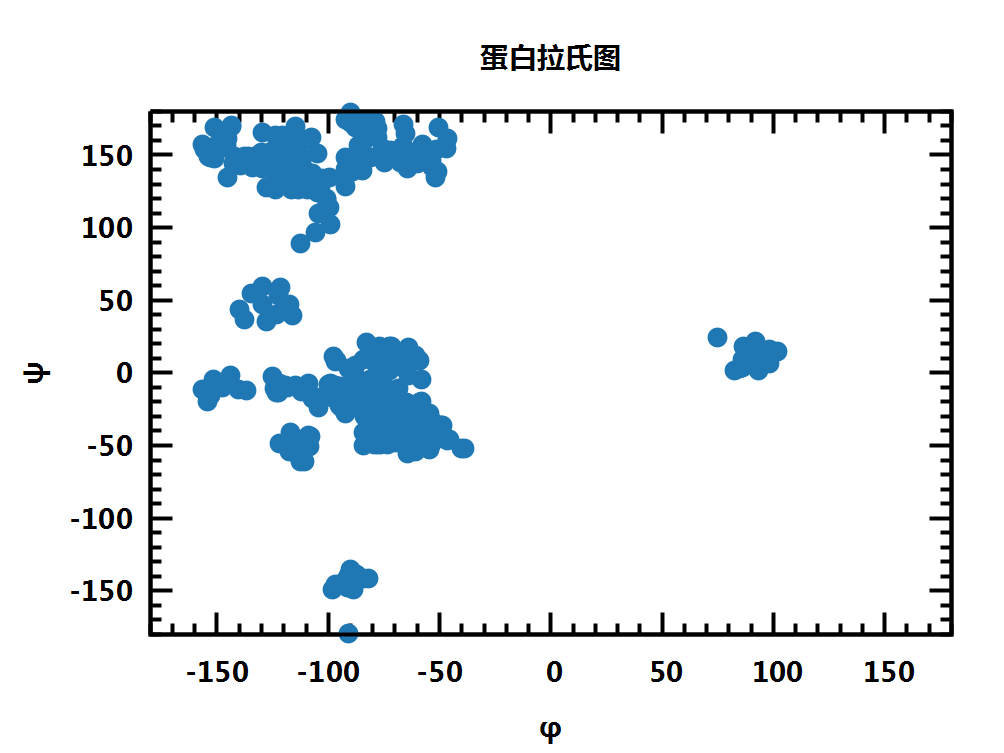
但点数多了之后, 就会重叠在一起, 很难分清, 不直观, 丢失了很多信息.
我们可以做得更好些, 在绘制每个点的时候设置一定的透明度, 这样点重叠越多的地方颜色就越深, 从而能够表明分布密度的大小.
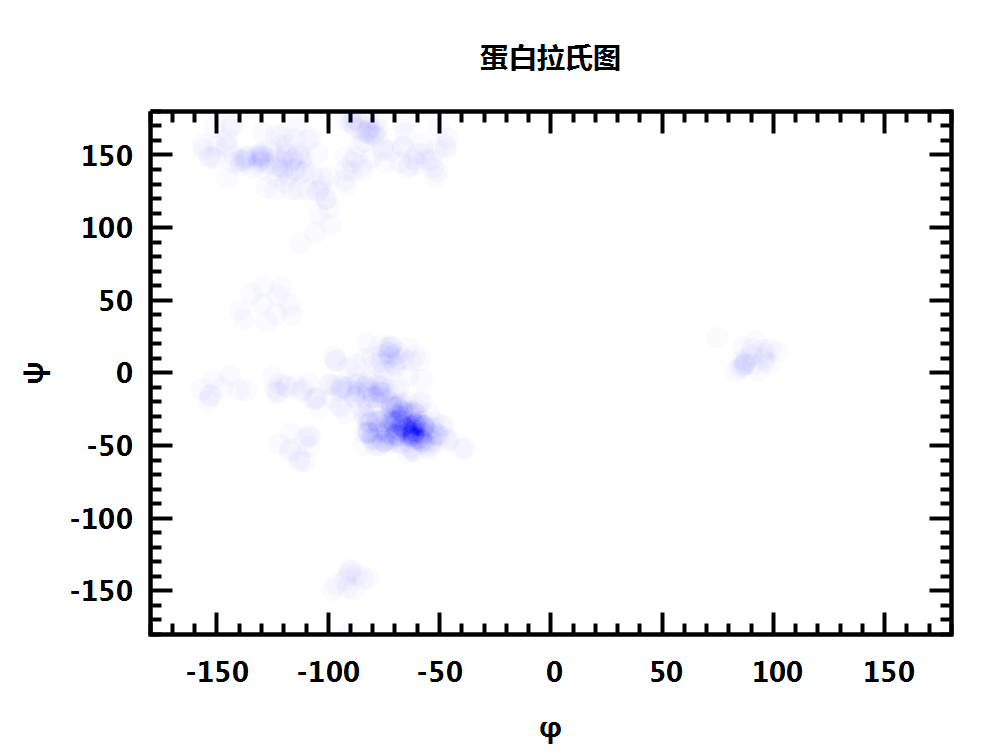
绘制这种图虽然简单, 但透明度很难提前预知, 需要测试才知道什么值给出的效果最好.
最好的解决方案还是绘制热图heatmap. 但这需要先统计(φ,ψ)的二维频数. 简单的几句脚本就可以解决这个问题, 但我还是希望能直接在gnuplot中解决, 所以就花了一点时间看看到底能不能做成. 想了几种貌似可行的简洁方案, 但实际证明都不可行, 因为gnuplot脚本的编程功能有很多限制, 最终还是先计算出频数, 再绘制. 计算时, 使用stat的统计功能, 直接累加处于一定(φ,ψ)范围内的数据个数即可. 方法很笨, 效率很低, 暂时还没想到更优雅的.
| gnuplot | |
|---|---|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 | eval setpal('cm_parula')
dphi=2
dpsi=2
set print $rama
do for [phi=-180:180:dphi] {
do for [psi=-180:180:dpsi] {
stat 'rama.xvg' u ( (phi-dphi/2<=$1 && $1<phi+dphi/2 && psi-dpsi/2<=$2 && $2<psi+dpsi/2) ? 1:0) nooutput
print phi, psi, STATS_sum
}}
set tit"蛋白拉氏图"
set xl"φ"; set yl"ψ"
set label "α螺旋(3.6_{13})" at -57.8, -47 front font ",50" textcolor "white"
set label "β折叠(反平行)" at -139, 135 front font ",50" textcolor "white"
set label "β片(平行)" at -119 , 113 front font ",50" textcolor "white"
set label "π螺旋(4.4_{16})" at -57.1, -69.7 front font ",50" textcolor "white"
set label "γ螺旋(2.2_7)" at -70 , 70 front font ",50" textcolor "white"
set label "2.0_5螺旋" at -175 , -175 front font ",50" textcolor "white"
set label "3.0_{10}螺旋" at -49 , -26 front font ",50" textcolor "white"
set label "PPI 螺旋" at -75 , 160 front font ",50" textcolor "white"
set label "PPII 螺旋" at -75 , 150 front font ",50" textcolor "white"
set label "左手α螺旋" at 57.8 , 47 front font ",50" textcolor "white"
plot [-180:180][-180:180] $rama u 1:2:3 w imag |
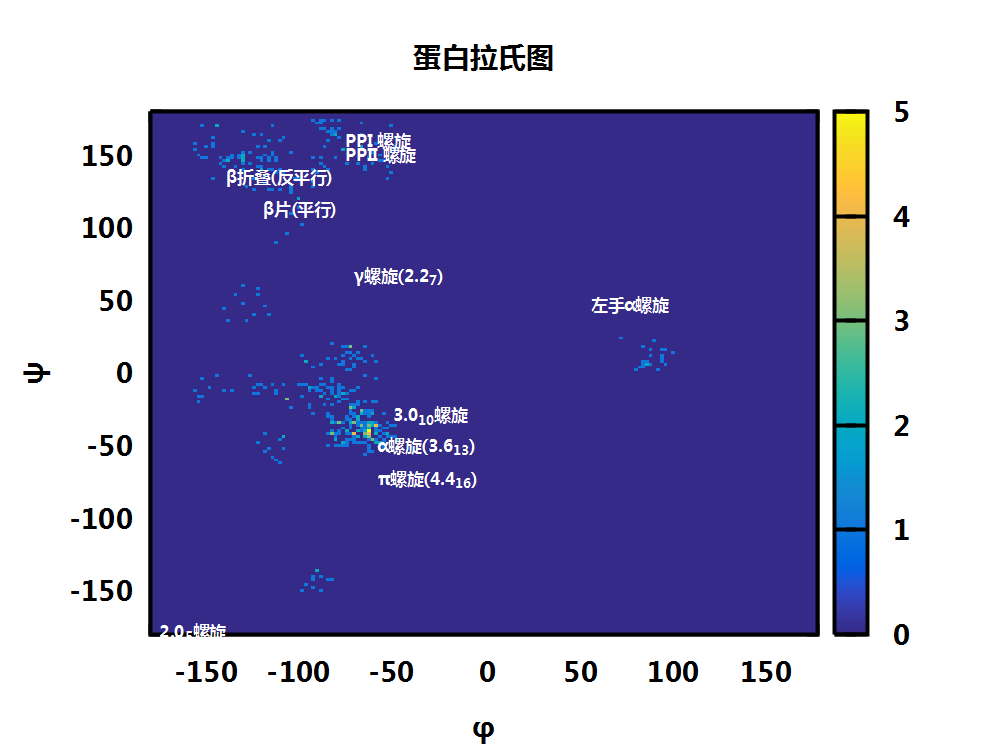
最后说一句, python的matplotlib有直接绘制2D直方图的功能, 可以参考Visualization with Matplotlib. 这本书有中文版, 值得推荐.
