- 2024-04-02 14:47:55
【案】在参加某个 workshop 的时候用到了这种算法及其 python 代码, 简单读了一下相关的论文, “Geodesic Interpolation For Reaction Pathways”; Xiaolei Zhu, Keiran C. Thompson, Todd J. Martínez; J. Chem. Phys. 150(16):164103, 2019; https://dx.doi.org/10.1063/1.5090303, 涉及微分几何, 黎曼流形, 度规张量, 曲率联络等, 广义相对论的感觉, 也不大懂, 好在代码用起来简单, 也就不求甚解直接用了. 现将其 GitHub 的 README 翻译整理出来, 供参考. 翻译草稿来自 ChatGPT-4, 并稍加修订, 确然无甚功劳可言, 旨在方便中文母语者速览知其大意.
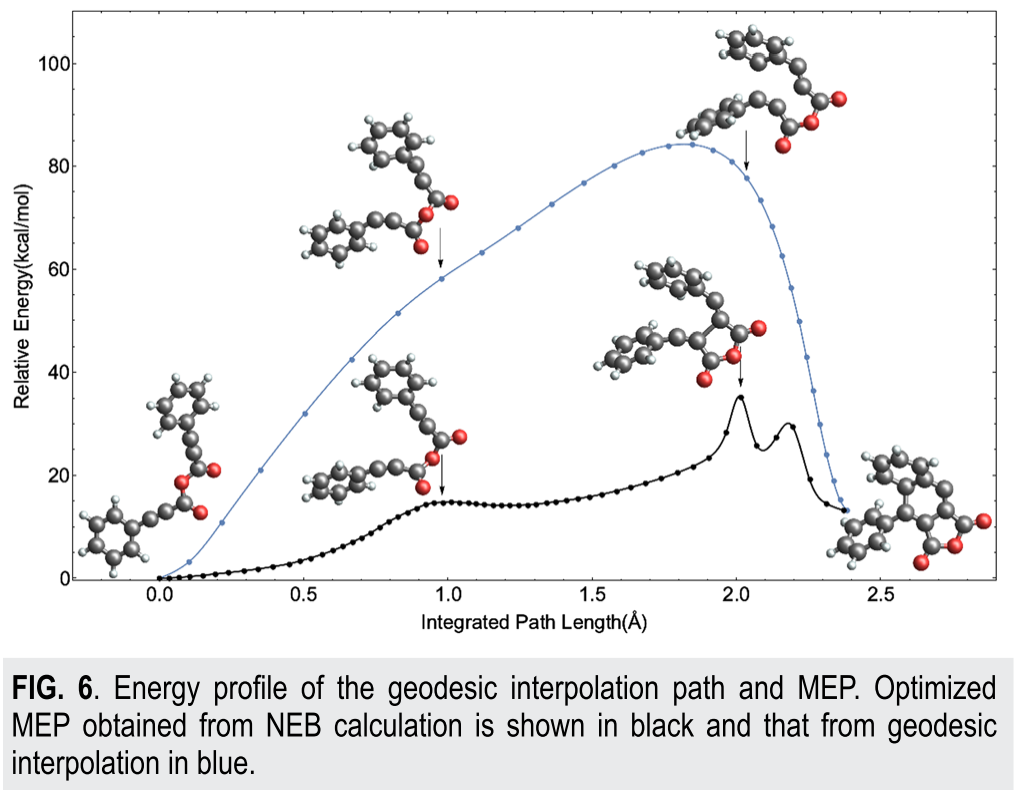
利用测地插值算法, 可以在分子几何构型之间进行插值, 构建插值路径, 以其作为反应途径.
在处理冗余内坐标空间时, 传统的插值方法会遇到困难, 因为可行的物理空间是一个非常小, 且高度曲折的子空间. 本方法通过严格在可行空间内操作避免了可行性问题, 并通过恰当使用对应的度规张量体现了内坐标的优势. 使用这种新的思路, 我们将构型空间视为一个黎曼流形, 其度规由一组内坐标生成, 插值路径定义为此类流形上的测地曲线, 换句话说, 总坐标变化的积分是最小的. 这样的定义确保了构建的路径平滑且行为良好. 该软件包也可用于平滑来自 MD 模拟的不连续或带噪声轨迹.
研究表明, 即使对于高度复杂的反应, 如蛋白质去折叠, 或伴随许多环同时形成的协同环加成反应, 该方法也能生成具有合理势垒高度的平滑路径. 默认坐标系使用Morse缩放的成对距离, 此坐标系中的长度具有物理意义, 表征了沿路径键变化的总次数.
脚本
本软件包是一个纯Python实现, 因此没有优化运行速度, 而是旨在作为所述算法的参考实现. 尽管如此, 插值包含约1000个原子的体系也不成问题.
geodesic_interpolate: Python包, 通过寻找具有冗余内部度规的测地曲线进行插值和平滑__init__.py: Python包文件__main__.py: 用于执行插值和平滑的独立脚本.geodesic.py: 在冗余内度规中计算和最小化路径长度. 用于优化路径以找到测地线, 在优化过程中不能改变构型数量interpolation.py: 生成近似插值点作为测地线优化的初始猜测. 尝试在内部空间中对最大段进行递归二分, 创建的路径通常不连续, 因此需要进行后续平滑处理.coord_utils.py: 坐标工具. 一个简化版的 Nanoreactor 坐标模块. 提供了缩放的原子间距离坐标、基于阈值和连接性的成对筛选, 以及基于Kabsch算法的平移-旋转对齐.fileio.py: XYZ文件的读取和写入setup.py: 安装脚本. 会安装Python包以及同独立脚本, 前者可通过geodesic_interpolate名称导入, 后者具有相同的名称.test_cases: 用于检查代码性能的一些测试案例. 请注意, 由于Python, 大型案例可能需要运行几分钟. 在测试另外的坐标缩放方法时尤其需要运行它们.
依赖
- Python : >2.7 或 >3.5
- Numpy : 测试版本为1.13
- Scipy : 测试版本为0.19
安装
不安装时可以在包目录中使用:
F:\Python3.8\python.exe -m geodesic_interpolate filename ...
要在任意位置使用脚本或使用Python模块, 请使用setup工具进行安装:
python setup.py install
这会安装一个Python包geodesic_interpolate和一个独立的脚本geodesic_interpolate. 安装后, 可以使用上述命令行从任意位置调用包, 也可以使用具有相同名称的独立脚本
geodesic_interpolate filename.xyz --output output.xyz --nimages 20 ...
使用
geodesic_interpolate [-h] [--nimages NIMAGES] [--sweep] [--no-sweep]
[--output OUTPUT] [--tol TOL] [--maxiter MAXITER]
[--microiter MICROITER] [--scaling SCALING]
[--friction FRICTION] [--dist-cutoff DIST_CUTOFF]
[--logging {DEBUG,INFO,WARNING,ERROR}]
[--save-raw SAVE_RAW]
filename
在两个几何构型之间进行插值
位置参数:
filename: 包含几何构型的XYZ文件. 如果构型数量小于所需数量, 会添加插值点; 如果数量过多, 会进行下采样.
可选参数:
-h.--help: 显示此帮助信息并退出--nimages NIMAGES: 构型数量. (默认:17)--sweep: 扫描路径. 一次优化一个构型, 而不是同时移动所有构型. 如果原子数量超过30, 则默认执行扫描更新. (默认:None)--no-sweep: 不进行扫描. (默认:None)--output OUTPUT: 输出文件名. 默认为interp.xyz(默认:interpolated.xyz)--tol TOL: 收敛容差(默认:0.002)--maxiter MAXITER: 最小化迭代的最大次数(默认:15)--microiter MICROITER: 扫描算法的最大微迭代次数. (默认:20)--scaling SCALING: Morse势的指数参数(默认:1.7)--friction FRICTION: 用于防止几何构型变化过大的摩擦项大小. (默认:0.01)--dist-cutoff DIST_CUTOFF: 原子对之间距离的截止值, 用于决定是否将原子对包含在坐标系中. (默认:3)--logging {DEBUG,INFO,WARNING,ERROR}: 日志级别[ DEBUG | INFO | WARNING | ERROR ](默认:INFO)--save-raw SAVE_RAW: 指定后, 保存二分之后且平滑之前的原始路径. (默认:None)
示例
test_cases目录下有几个示例文件及其输出.
H+CH4_CH3+H2.xyz: 简单测试
一个简单的测试案例, 应该不存在问题.
DielsAlder.xyz: Diels-Alder脱氢反应
这是一个重要的测试案例, 因为初始结构具有平面对称性, 最终结构及其镜像都可以达到, 二者具有完全相同的内坐标. 因此, 插值过程中正确监测测地长度至关重要, 否则, 原始插值路径会在镜像之间跳跃, 优化后的路径会包含一些反折. 还测试了在相对较大的体系中非扫描全局路径优化算法.
TrpCage_unfold.xyz: Trp-cage微蛋白的去折叠.
至少需要10个构型. 折叠几何结构取自Stefan的测试目录, 源于Nathan. 去折叠结构来自1 ns的力导向MD, 力场为ReaxFF, 1 nN的力施加在C和N末端, 所得结构在B3LYP/6-31g水平进行了优化, 且优化时未考虑外力. 用于测试许多同时发生的大幅度运动.
collagen.xyz: 胶原蛋白
在胶原蛋白Kunitz域1kun - 1kth 的溶液和晶体结构之间进行插值. 移除了PDB结构中的溶剂. 测试避免折叠蛋白核心的大幅度移动导致的碰撞. 其他组应该稍微松弛以便为变化的部分留出空间.
calcium_binding.xyz: 酵母钙调蛋白
将两个Ca2+离子结合到酵母钙调蛋白N末端域1f54 -1f55. apo结构无离子, 因此在远离蛋白的位置手动添加了两个离子. 当Ca2+阳离子离蛋白质非常远时, 似乎很难避免它们出现大幅度移动, 但一旦接触到蛋白, 它们应该能够平滑移动. 这是为了测试插值算法是否能正确规划路径, 找到入口点并连接路径.
