原文更新于 Friday, 19 August 2016 13:07
发布于2016-08-31 15:26:13 翻译: 黄建湘; 校对: 李继存
Martini力场是一种适用于生物分子体系分子动力学模拟的粗粒化(CG, coarse-grain)力场, 该力场采用系统化的方式进行参数化, 结合了”自上而下”和”自下而上”两种拟合策略: 非键相互作用基于再现大量化合物在极性和非极性溶剂中分配自由能的实验值, 而键合相互作用则来源于参考的全原子模拟.
Martini模型采用四合一的映射方式, 即平均来说, 将使用一个作用中心来代表四个重原子及与其连接的氢原子. 为了保持模型简洁, 只定义了四种主要的相互作用位点类型: polar(极性), non-polar(非极性, 亲水), apolar(无极性, 疏水), charged(荷电). 每种粒子类型可进一步划分为几种亚型, 这样可以准确地代表分子原子结构的化学本质.
non-polar和apolar的区别可理解为极性/亲疏水性(polarity/hydrophilicity), 使用中表现为Lennard-Jones参数不同.
- apolar: C-type, 具有很强的疏水性
- non-polar: N-type, 可溶于水和有机溶剂
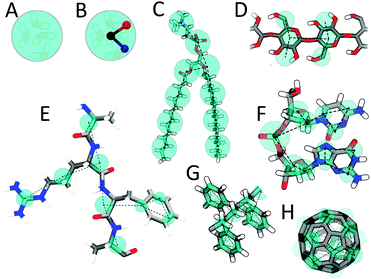
分子Martini映射的一些例子: (A) 代表了四个水分子的标准水粒子; (B) 嵌入电荷的可极化水分子; (C) DMPC磷脂; (D) 多糖片段; (E) 多肽; (F) DNA片段; (G) 聚苯乙烯片段; (H) 富勒烯分子. 在所有例子中Martini CG珠子都显示为覆盖在原子结构外面的青色透明珠子.
目前有许多磷脂分子和表面活性剂分子的拓扑可以使用, 包括胆固醇, 所有的氨基酸, 一系列的糖, 聚合物和纳米粒子. 另外, 还可以进一步通过脚本来构建任意多肽和蛋白质的拓扑, 增加弹性网络(elastic network), 在粗粒化表示和原子表示之间转换. 有关Martini模型的细节可以在如下出版物中找到:
- S.J. Marrink, D.P. Tieleman. Perspective on the Martini model. Chem. Soc. Rev., 42:6801-6822, 2013. 开放获取
- D.H. de Jong, G. Singh, W.F.D. Bennett, C. Arnarez, T.A. Wassenaar, L.V. Schafer, X. Periole, D.P. Tieleman, S.J. Marrink. Improved parameters for the Martini coarse-grained protein force field, JCTC, 9:687-697, 2013. 开放获取
- X. Periole, S.J. Marrink. The Martini coarse-grained force field. In “Methods in molecular biology”, Vol 924, L. Monticelli & E. Salonen Eds., Springer, 2013, pp 533-565. 摘要, 重印本
- S.O. Yesylevskyy, L.V. Schäfer, D. Sengupta, S.J. Marrink. Polarizable water model for the coarse-grained Martini force field. PLoS Comp. Biol, 6:e1000810, 2010. 开放获取
- C.A. Lopez, A. Rzepiela, A.H. de Vries, L. Dijkhuizen, P.H. Huenenberger, S.J. Marrink. The Martini coarse grained force field: extension to carbohydrates. J. Chem. Theo. Comp., 5:3195-3210, 2009. 摘要
- S.J. Marrink, M. Fuhrmans, H.J. Risselada, X. Periole.The MARTINI force field. In “Coarse graining of condensed phase and biomolecular systems”, G. Voth ed., CRC press, Chapter 2, 2008.
- L. Monticelli, S.K. Kandasamy, X. Periole, R.G. Larson, D.P. Tieleman, S.J. Marrink. The MARTINI coarse grained forcefield: extension to proteins. JCTC, 4:819-834, 2008.
- S.J. Marrink, H.J. Risselada, S. Yefimov, D.P. Tieleman, A.H. de Vries. The MARTINI forcefield: coarse grained model for biomolecular simulations. JPC-B, 111:7812-7824, 2007.
- S.J. Marrink, A.H. de Vries, A.E. Mark. Coarse grained model for semi-quantitative lipid simulations. JPC-B, 108:750-760, 2004.
