- 2017年04月03日 20:43:00
【按】以下翻译自Amber 16手册.
13.6 构建寡糖, 脂和糖蛋白
GLYCAM网站支持的功能
GLYCAM网站可以自动生成下面的分子, 且支持许多附加选项. 该网站的功能正在扩展中, 目前可用的功能包括:
- 寡糖, 线性和枝状的
- 糖蛋白, 与O或N相连的, 含有多个糖单元
- 通过URL构建寡糖
AmberTools测试提供的辅助
除后面的示例外, AmberTools测试文件中还包含了其他示例. 相关文件位于
$AMBERHOME/AmberTools/test/LEaP/GLYCAM: 测试主目录$AMBERHOME/AmberTools/test/LEaP/GLYCAM/06EPb: 附加位点的寡糖$AMBERHOME/AmberTools/test/LEaP/GLYCAM/06j: 寡糖主目录$AMBERHOME/AmberTools/test/LEaP/GLYCAM/06j_10: 与ff10力场联用的糖蛋白$AMBERHOME/AmberTools/test/LEaP/GLYCAM/06j_12SB: 与ff12SB力场联用的糖蛋白
注意 对于测试目录中的分子, 其构建目的仅在于测试AmberTools, 可能并不能直接用于模拟. 有些构象可能不合理, 原子间距过小. 大多数结构问题都可以通过适当修改扭转角来解决. 在糖蛋白测试文件中, 包含了利用impose命令控制扭转角的使用示例.
GLYCAM下的每一子目录包含了与特定力场相关的测试. 要运行某个测试, 先保存所有相关的输出文件和中间文件, 然后切换到子目录, 运行下面的命令
./Run.GLYCAM evaluate
要将目录还原为初始状态, 运行
./Run.GLYCAM clean
主目录中的00_README文件包含了有关测试的更多信息.
附加说明
继续本节前, 你应该先学习3.3节中有关GLYCAM的命名约定. 之后, 还要记住两件重要的事情. 第一件是, GLYCAM设计用于构建寡糖, 而不仅是单糖. 为了将单糖连接起来, GLYCAM的每一残基必须至少含有一个不饱和位置, 也就是说, 每一个GLYCAM残基, 或者缺少一个羟基, 或者缺少一个羟基氢原子, 依赖于分枝位置的数量, 也可能缺少多个氢原子. 因此, 每个残基自身并不是一个完整的分子. 例如, 如果你要构建α-D-型吡喃葡萄糖, 你必须明确地指定端基异构的-OH官能团(参见图13.1中的两个例子).
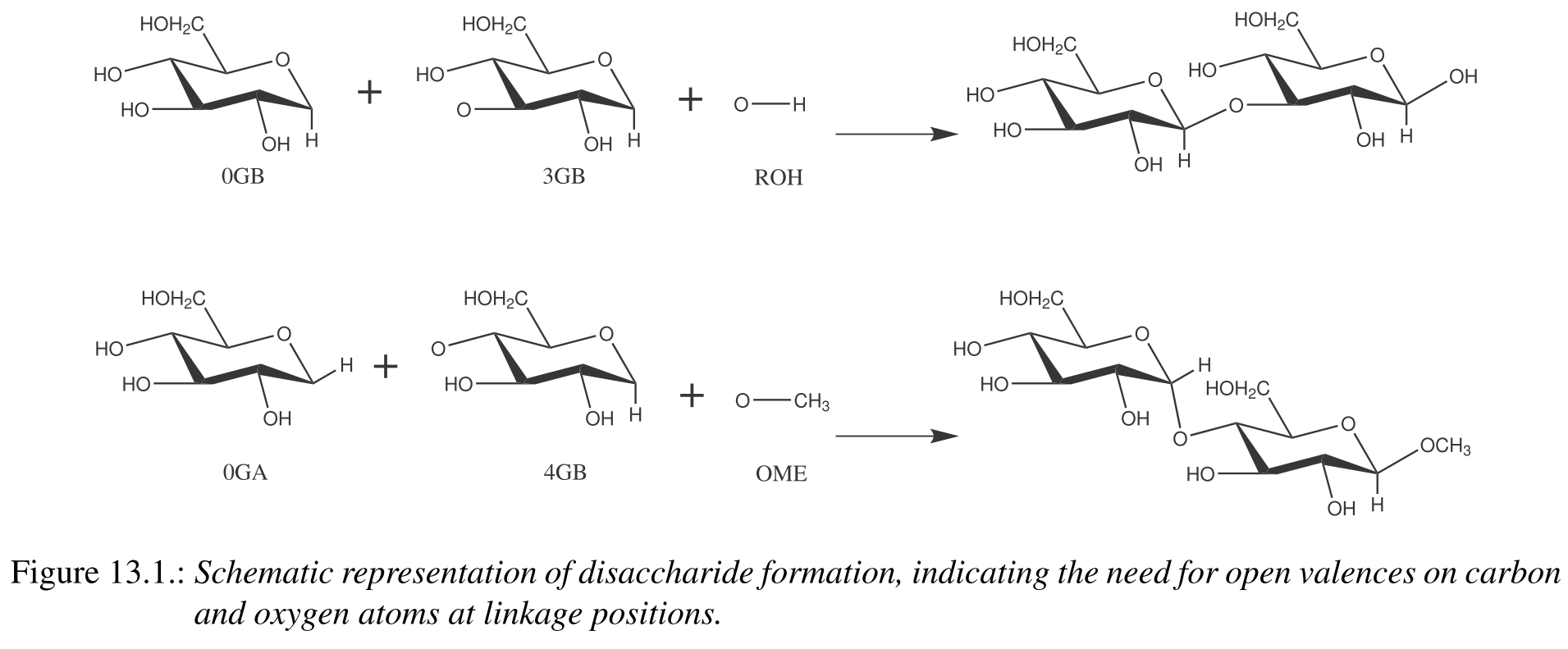
第二件要记住的是, 当在LEaP中使用sequence命令来连接单糖形成线性寡糖(类似于生成多肽)时, 残基顺序与书写残基序列的标准约定顺序相反.
例如, 要构建图13.1中的二糖, 使用LEaP的sequence命令, 格式为
upperdisacc = sequence { ROH 3GB 0GB }
lowerdisacc = sequence { OME 4GB 0GA }
尽管sequence命令是构建线性多糖最直接的方法, 但并非唯一的方法. 下面会介绍构建更复杂多糖和糖蛋白的其他方法. 如果要构建更复杂的结构(并创建拓扑和坐标文件), 一种很方便的方法是使用GLYCAM在线工具http://glycam.ccrc.uga.edu或http://www.glycam.org.
本节中, LEaP命令以下面的格式给出(注意, 此格式不能用于sleap, 因其不支持注释和语句处于同一行.)
command argument(s) # 注释
之所以选择此格式是因为这样一行命令可以直接复制粘贴到文件中, 然后使用LEaP读取. #后的内容为注释, 以便将来参考, 执行时会被LEaP忽略. 要使用LEaP读入文件, 或者source文件, 或者在命令行中指定
tleap -f leap_input_file
注意, Amber自带的每一GLYCAM数据集未来都可能更新. 目前所带的版本为GLYCAM_06j.dat, 默认的leaprc.GLYCAM_06j-1会自动加载此文件和GLYCAM_06j-1.prep. 建议用户查看
13.6.1 使用GLYCAM-06参数构建寡糖的步骤
13.6.1.1 示例: 线性寡糖
本节示例如何创建一个简单的直链四糖
α-D-Manp-(1-3)-β-D-Manp-(1-4)-β-D-GlcpNAc-(1-4)-β-D-GlcpNAc-OH
首先要确定用于构建此寡糖的GLYCAM残基. 由于初始的α-D-Manp残基只在其端基异构位点连接, 其名称中的第一个字符为0(零), 表示它没有分枝或者其他连接, 即它是端基. 由于它是D型甘露糖, 第二个单字符编码为M(大写). 由于它是α吡喃糖, 第三个字符为A. 因此, 上面序列中的第一个残基为0MA. 由于第二个残基的连接点位于3号位置以及端基异构位置, 其名称的第一个字符为3, 作为β吡喃糖, 其名称为3MB. 类似的, 第三个和第四个残基都是4YB. 要创建完整的分子, 需要在末端添加OH残基. 注意在下面的13.6节中, 端基OH 必须 忽略, 这样分子才能继续连接到蛋白或脂. 还要注意, 存在的末端OH(或OME等)残基会被赋予自己的残基编号.
将序列顺序反转一下以便用于LEaP的sequence命令:
残基名称序列: ROH 4YB 4YB 3MB 0MA
残基编号: 1 2 3 4 5
下面的LEaP指令用于构建上面的序列(当然, 也可以使用其他方法来做)
source leaprc.GLYCAM_06j-1 # 载入 leaprc
glycan = sequence { ROH 4YB 4YB 3MB 0MA } # 构建寡糖
使用sequence命令, φ角会自动设置, 其取向基于外端基异构效应的预计(±60°). 如果你要改变两个残基之间的扭转角, 可以使用impose命令. 在下面的例子中, 两个4YB残基之间, 4YB和3MB之间的ψ角被设置为标准值零.
impose glycan {3} { {C1 O4 C4 H4 0.0} } # 设定 4YB (3) 和 4YB (2) 之间的psi
impose glycan {4} { {C1 O4 C4 H4 0.0} } # 设定 3MB (4) 和 4YB (3) 之间的psi
现在你可以生成坐标, 拓扑以及pdb文件:
saveamberparm glycan glycan.top glycan.crd # 保存 top 和 crd
savepdb glycan glycan.pdb # 保存 pdb 文件
13.6.1.2 示例: 枝状寡糖
本节示例如何构建简单的枝状寡糖, 所用示例基于前一节构建的寡糖. 同样, 这里仍然假定要构建的糖分子并不与蛋白或脂相连. 否则, 你应该忽略结构中的ROH残基. 要构建的枝状寡糖为
α-D-Manp-(1-3)-β-D-Manp-(1-4)-β-D-GlcpNAc-(1-4)-β-D-GlcpNAc-OH
6
|
α-D-Manp-1
注意到, β-D-甘露吡喃糖的枝接位置在3和6号. 查看表格3.5到3.8, 对于3号和6号位置存在连接性的单糖, 其名称的第一个字符是V, 因此, 前一节中的残基名称3MV必须改为VMB.
重写用于此糖分子的LEaP命令
残基名称序列: ROH 4YB 4YB VMB 0MA 0MA
残基编号: 1 2 3 4 5 6
为保证正确的残基连接到VMB的3号和6号位置, 最保险的做法是在LEaP中明确地指定这些连接. 在当前的示例中, 两个末端残基是相同的(0MB), 但其他情况未必如此.
source leaprc.GLYCAM_06j-1 # 载入 leaprc
glycan = sequence { ROH 4YB 4YB VMB } # 待分枝的线性序列
首先构建最长的线性序列, 在分枝点VMB处结束, 以便明确地指定亚序列的连接性. 下面的命令会在3号位置放置一个末端残基0MB:
set glycan tail glycan.4.O3 # 将附加点设置为 VMB 中的 O3
glycan = sequence { glycan 0MA } # 添加其中一个 0MA
下面的命令将另一个0MA连接到6号位置. 注意, 分子名称由glycan变为branch. 此变化并非必要, 但能够使命令更易读, 尤其当构建复杂结构时.
set glycan tail glycan.4.O6 # 将附加点设置为 VMB 中的 O6
branch = sequence { glycan 0MA } # 添加另一个 0MA
当构建枝状寡糖时, 重新设定扭转角尤其重要. 下面的命令可以比较好地整理分子构型, 然后生成一系列输出文件:
impose branch {4} { {H1 C1 O6 C6 -60.0} } # 设定 phi 扭转角以及
impose branch {4} { {C1 O6 C6 H6 0.0} } # 设定 psi 0MA(6) & VMB
impose branch {4} { {H1 C1 O4 C4 60.0} } # 设定 phi 扭转角以及
impose branch {4} { {C1 O4 C4 H4 0.0} } # 设定 psi 3MB & 4YB
impose branch {3} { {H1 C1 O4 C4 60.0} } # 设定 phi 扭转角以及
impose branch {3} { {C1 O4 C4 H4 0.0} } # 设定 psi 4YB & 4YB
impose branch {5} { {H1 C1 O3 C3 -60.0} } # 设定 phi 扭转角以及
impose branch {5} { {C1 O3 C3 H3 0.0} } # 设定 psi 0MA(3) & VMB
saveamberparm branch branch.top branch.crd # 保存 top & crd
savepdb branch branch.pdb # 保存 pdb
13.6.1.3 示例: 复杂的枝状寡糖
下面的示例构建高度分枝, 高甘露糖比例的结构, 如图13.2所示. 在这个例子中, 尤其要说明的是, 当分枝容易模糊时, LEaP可能无法选择用户需要/预期的连接点. 为此, 无论何时结构分枝, 都必须明确地指定连接性. 也就是说, 不能先指定最长的线性序列, 然后加入分枝. sequence命令必须在每一分枝点进行, 否则, 并不能保证连接性正确. 在此例中, 分枝位于每个VMA(-3,6-D-Manp)残基中.
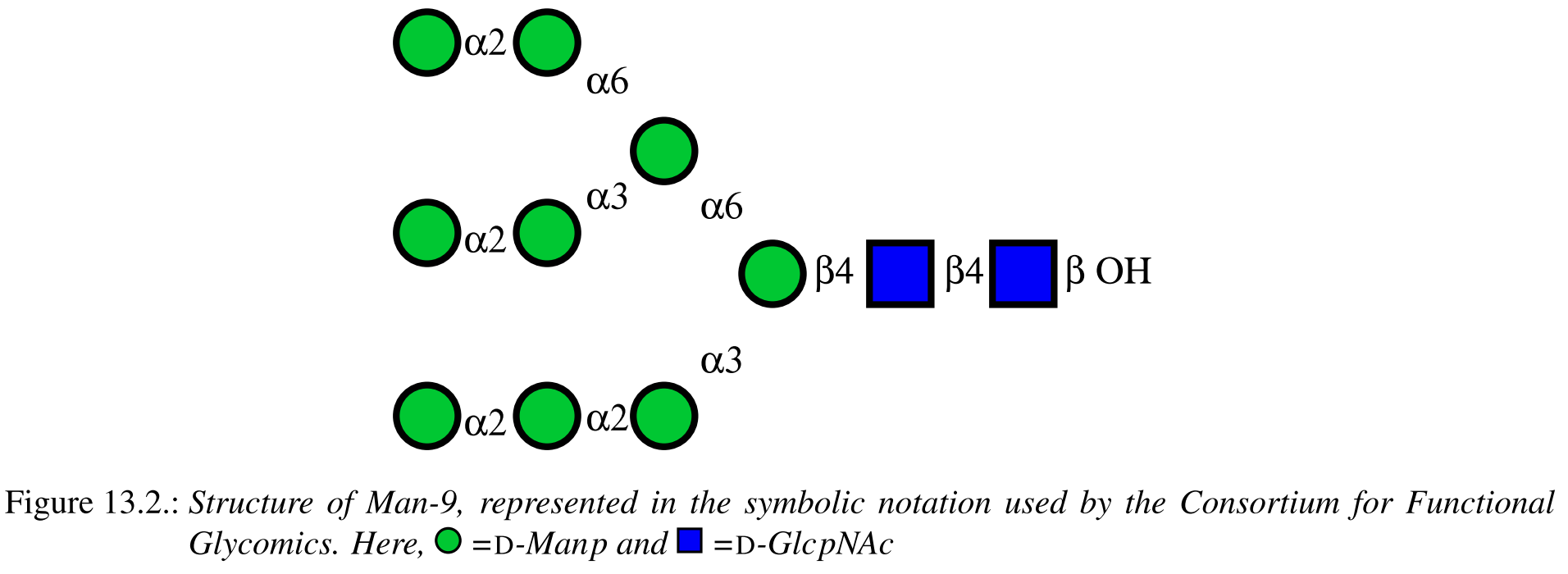
下面的tleap命令可以正确地生成图13.2中的结构
source leaprc.GLYCAM_06j-1
glycan = sequence { ROH 4YB 4YB VMB }
set glycan tail glycan.4.O6
glycan=sequence { glycan VMA }
set glycan tail glycan.5.O6
glycan=sequence { glycan 2MA 0MA }
set glycan tail glycan.5.O3
glycan=sequence { glycan 2MA 0MA }
set glycan tail glycan.4.O3
glycan=sequence { glycan 2MA 2MA 0MA }
impose glycan {3} { {H1 C1 O4 C4 60.0} }
impose glycan {3} { {C1 O4 C4 H4 0.0} }
impose glycan {4} { {H1 C1 O4 C4 60.0} }
impose glycan {4} { {C1 O4 C4 H4 0.0} }
impose glycan {5} { {H1 C1 O6 C6 -60.0} } # 1-6 Link from (5) to (4), Phi
impose glycan {5} { {C1 O6 C6 C5 180.0} } # 1-6 Link from (5) to (4), Psi
impose glycan {4} { {O6 C6 C5 O5 60.0} } # 1-6 Link from (5) to (4), Chi
impose glycan {10} { {H1 C1 O3 C3 -60.0} }
impose glycan {10} { {C1 O3 C3 H3 0.0} }
impose glycan {6} { {H1 C1 O6 C6 -60.0} }
impose glycan {6} { {C1 O6 C6 C5 180.0} }
impose glycan {5} { {O6 C6 C5 O5 -60.0} }
impose glycan {8} { {H1 C1 O3 C3 -60.0} }
impose glycan {8} { {C1 O3 C3 H3 0.0} }
impose glycan {7} { {H1 C1 O2 C2 -60.0} }
impose glycan {7} { {C1 O2 C2 H2 0.0} }
impose glycan {9} { {H1 C1 O2 C2 -60.0} }
impose glycan {9} { {C1 O2 C2 H2 0.0} }
impose glycan {11} { {H1 C1 O2 C2 -60.0} }
impose glycan {11} { {C1 O2 C2 H2 0.0} }
impose glycan {12} { {H1 C1 O2 C2 -60.0} }
impose glycan {12} { {C1 O2 C2 H2 0.0} }
saveamberparm glycan glycan.prmtop glycan.restrt
