- 2017年04月05日 19:23:46
【按】以下翻译自Amber 16手册.
13.6.2 使用GLYCAM-06参数构建脂分子
遵循这里讲述的步骤, 用户可以得到一个简单的脂分子, 但在构建过程中我们没有考虑它的轴向排列情况. 脂双层通常放置在直角坐标系统的(x, y)平面内, 这要求每个脂分子以从亲水”头”端到憎水”尾”端的顺序沿z方向排列. 通过载入已经沿z轴正确排列好的模板PDB文件, 这容易完成.
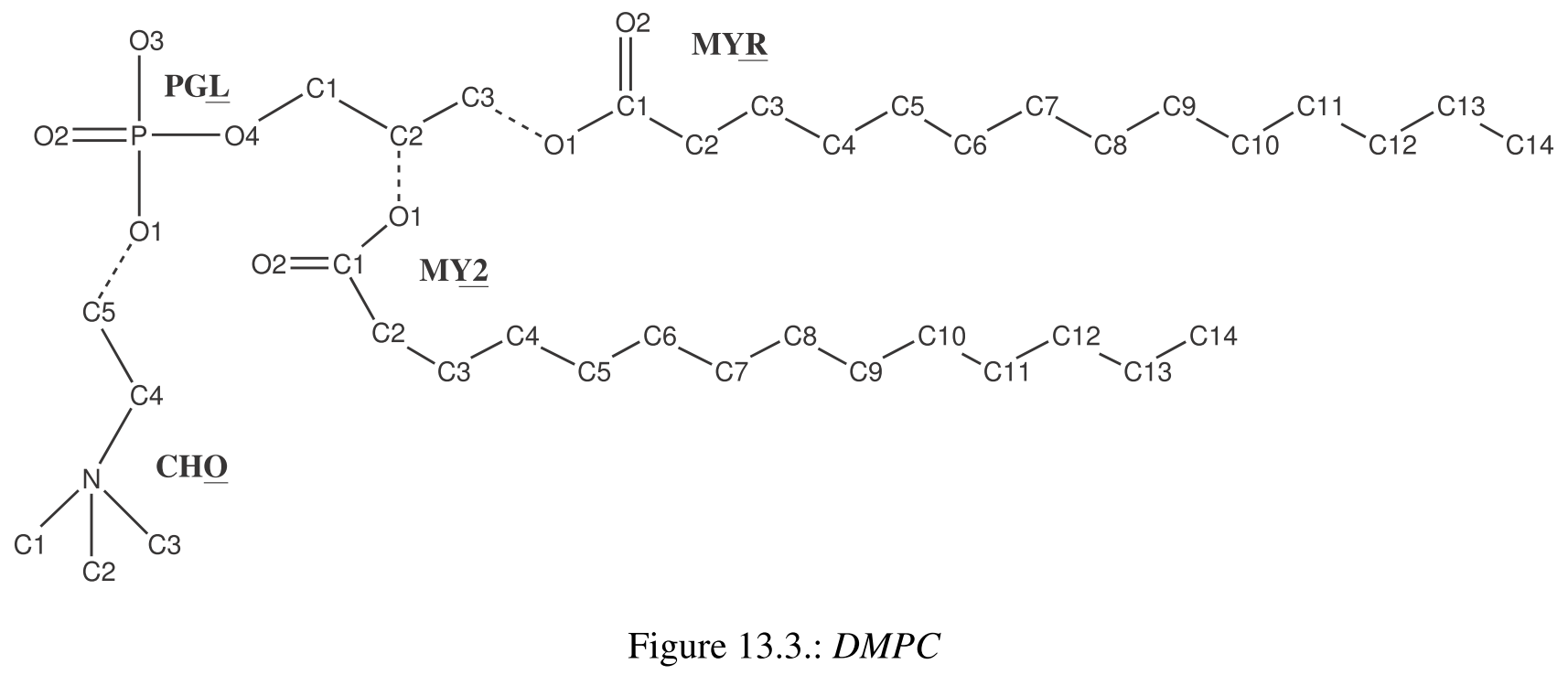
本例中我们使用的脂分子是二肉豆蔻酰磷脂酰胆碱(1,2-dimyristoyl-sn-glycero-3-phosphocholine), 或简称DMPC, 它由四个片段组成: 胆碱头基CHO, 磷酯头基PGL, sn-1链肉豆蔻酸尾基MYR, sn-2链肉豆蔻酸尾基MY2. 分子结构与原子编号如图13.3(为清晰起见, 未显示氢原子和原子电荷), 其中残基间的成键点以虚线表示. 本教程将只使用每个片段prep文件. 这些prep文件开始是pdb格式, 然后使用antechamber将其重新格式化为prep文件, 并添加了与GLYCAM兼容的电荷. prep文件数据库(GLYCAM_lipids_06h.prep)中已经包含了四个残基的相关文件.
13.6.2.1 示例: 使用LEaP构建脂分子
为了使用GLYCAM-06参数集构建一个脂分子, 你不需要加载整个GLYCAM pre文件, 但当使用默认的leaprc.GLYCAM_06j-1时它会自动载入. 注意, 使用下面的命令构建的脂分子还没有沿需要的方向对齐, 可能无法马上用于创建脂双层. 具体命令如下
source leaprc.GLYCAM_06j-1 # source GLYCAM-06的 leaprc 文件
loadamberprep GLYCAM_06_lipids.prep # 加载脂的 prep 文件
set CHO tail CHO.1.C5 # CHO的尾原子设为 C5
set PGL head PGL.1.O1 # PGL的头原子设为 O1
set PGL tail PGL.1.C3 # PGL的尾原子设为 C3
lipid = sequence { CHO PGL MYR } # 生成脂的直链部分
set lipid tail lipid.2.C2 # PGL的尾原子设为 C2
lipid = sequence { lipid MY2 } # 将 MY2 添加到 "lipid" 部分
impose lipid {2} { {C1 C2 C3 O1 163} } # 设定扭转角
impose lipid {2} { {C2 C3 O1 C1 -180} } # PGL & MYR
impose lipid {2} { {C3 O1 C1 C2 180} }
impose lipid {2} { {O4 C1 C2 O1 -60} } # 设定扭转角
impose lipid {2} { {C1 C2 O1 C1 -180} } # PGL & MY2
impose lipid {2} { {C2 O1 C1 C2 180} }
# 注意上面设定的扭转角数值可能不是最佳值
savepdb lipid DMPC.pdb # 保存 pdb 文件
saveamberparm lipid DMPC.top DMPC.crd # 保存 top 和 crd 文件
