- 2021-03-30 21:00:11
xpm2all脚本有段时间没有更新了, 因为没有看到有人提问题, 大约是用的人不多吧.
最近有人在绘制二级结构图的时候遇到了两个问题: 一个是对模拟时间较长的轨迹, 数据量很大, 两种绘制方法都很慢, 而且还不一定能绘制出来; 另一个问题是, 脚本中能处理的二级结构类型少了5螺旋(π螺旋), 导致对某些含5螺旋的蛋白出错.
第一个问题的原因在于, 原先的绘制方法是利用了gnuplot的自定义绘图功能, 相当于拿笔一点一点地画. 虽然这种方法自由度很高, 可以画任何东西, 但gnuplot并不是设计用来做这个的, 所以处理起来远远慢于直接绘制数据. 要想提高绘制速度, 可以直接使用类似热图的绘制方法, 方法可以参考以前的一篇文章使用gnuplot绘制xpm文件对应的数据.
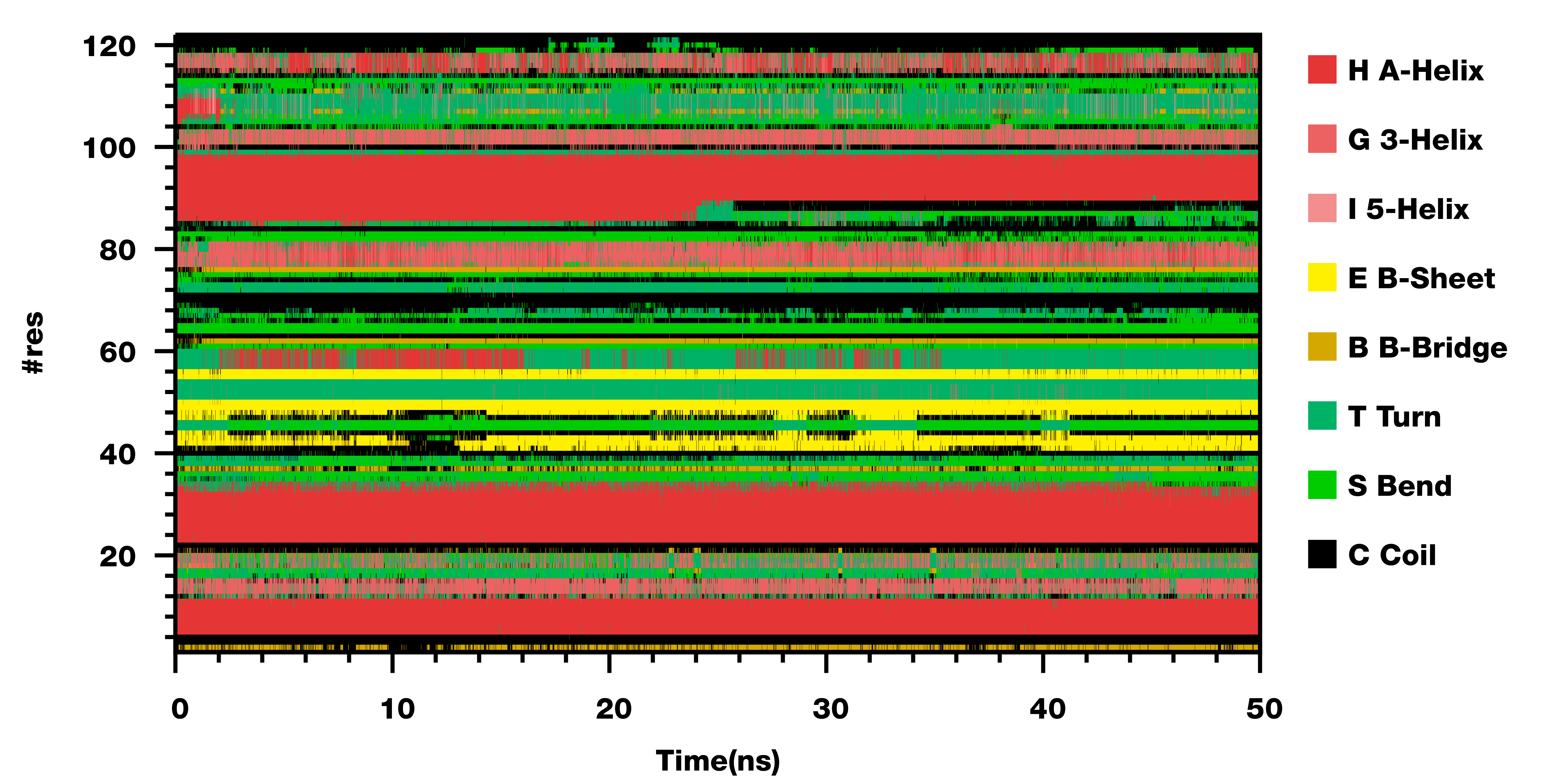
对模拟时间较长的轨迹, 运行gmx do_dssp时候建议使用-tu ns选项, 这样输出的ss.xpm中时间单位为ns, 绘图更好些. 下面是一个示例, 蛋白含122个残基, 不算大, 模拟了50 ns, 每2 ps保存一帧轨迹并计算二级结构. 就有点大了. 原先的脚本在处理这么大的数据量时很容易卡死, gnuplot绘制也很难成功. 使用新的方法, 绘制起来就没有什么压力了.
对第二个问题, 很简单, 为脚本新增加一个二级结构类型即可.
修订了这两个问题之后, 也顺便看了一下绘制二级结构时所用的颜色方案. 对于每种二级结构该用哪种颜色表示, 目前看起来并没有标准. GROMACS默认有一套颜色方案, VMD有一套, PDB也有一套, 文献上还有各种不同的颜色方案. 我试了试, 觉得都不怎么漂亮, 但也没有能力或精力自己设计一套, 所以也就只能给个选项, 把我测试过的加上. 如果你看到更好的颜色方案, 欢迎告知.
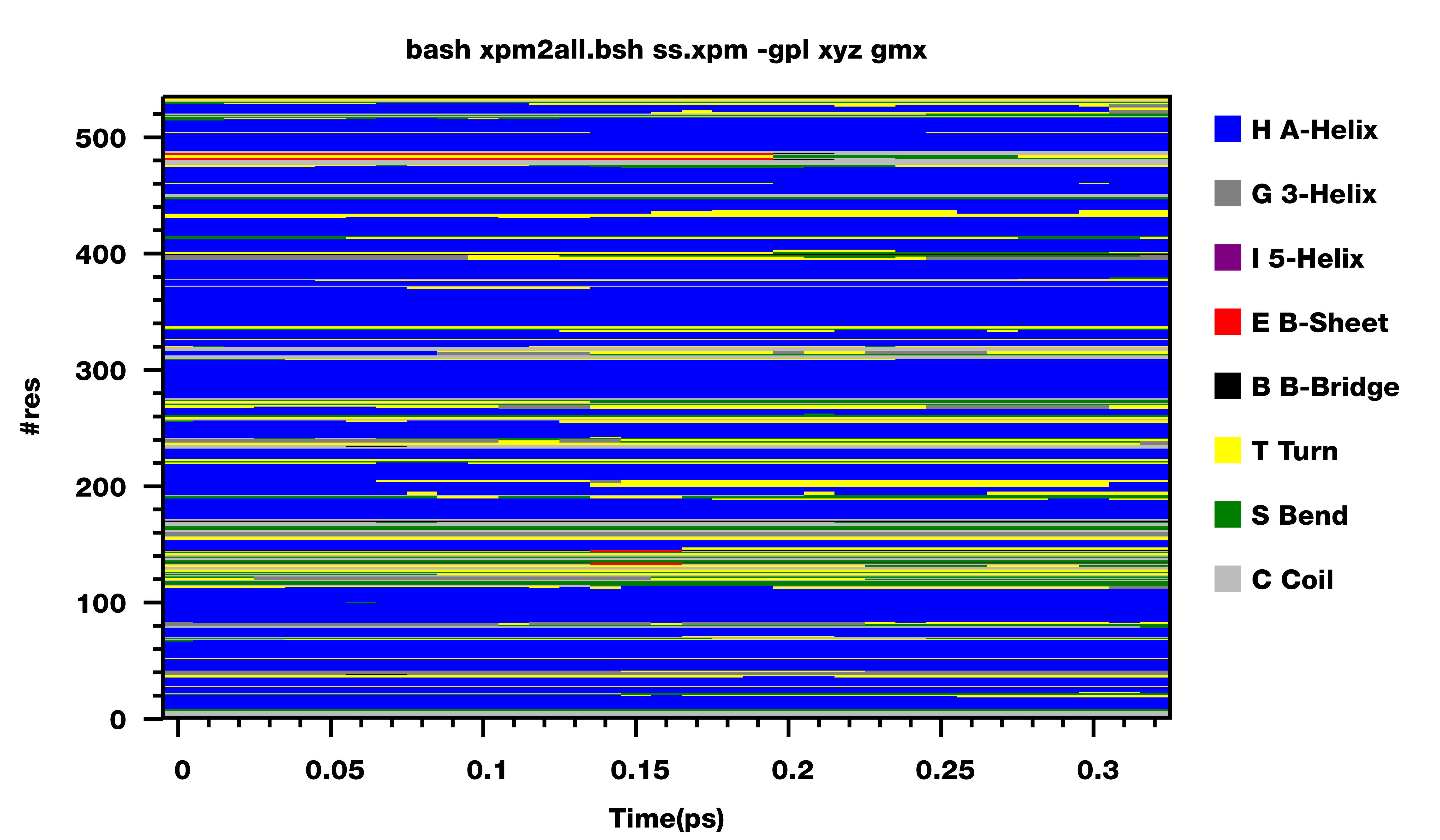
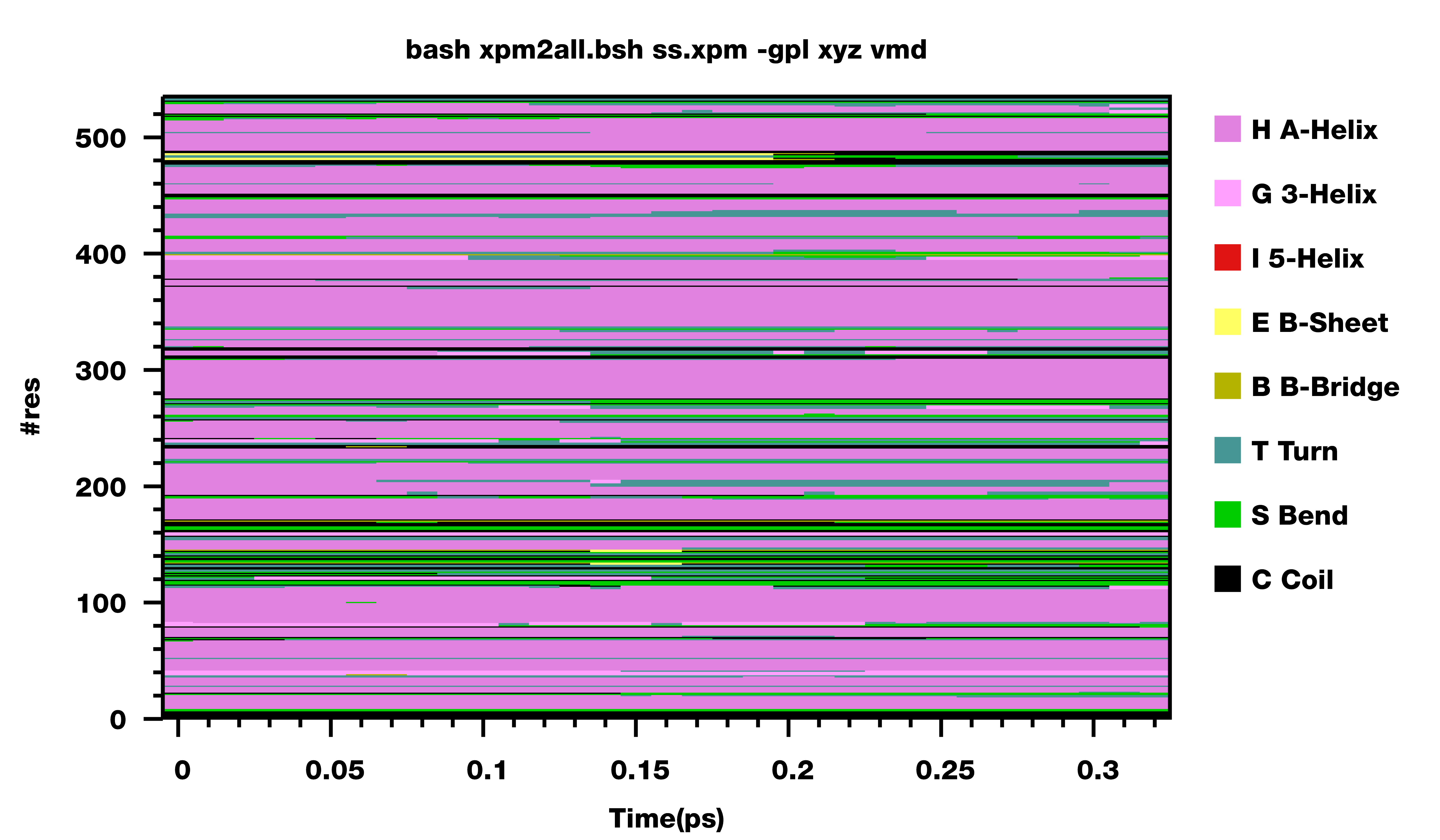
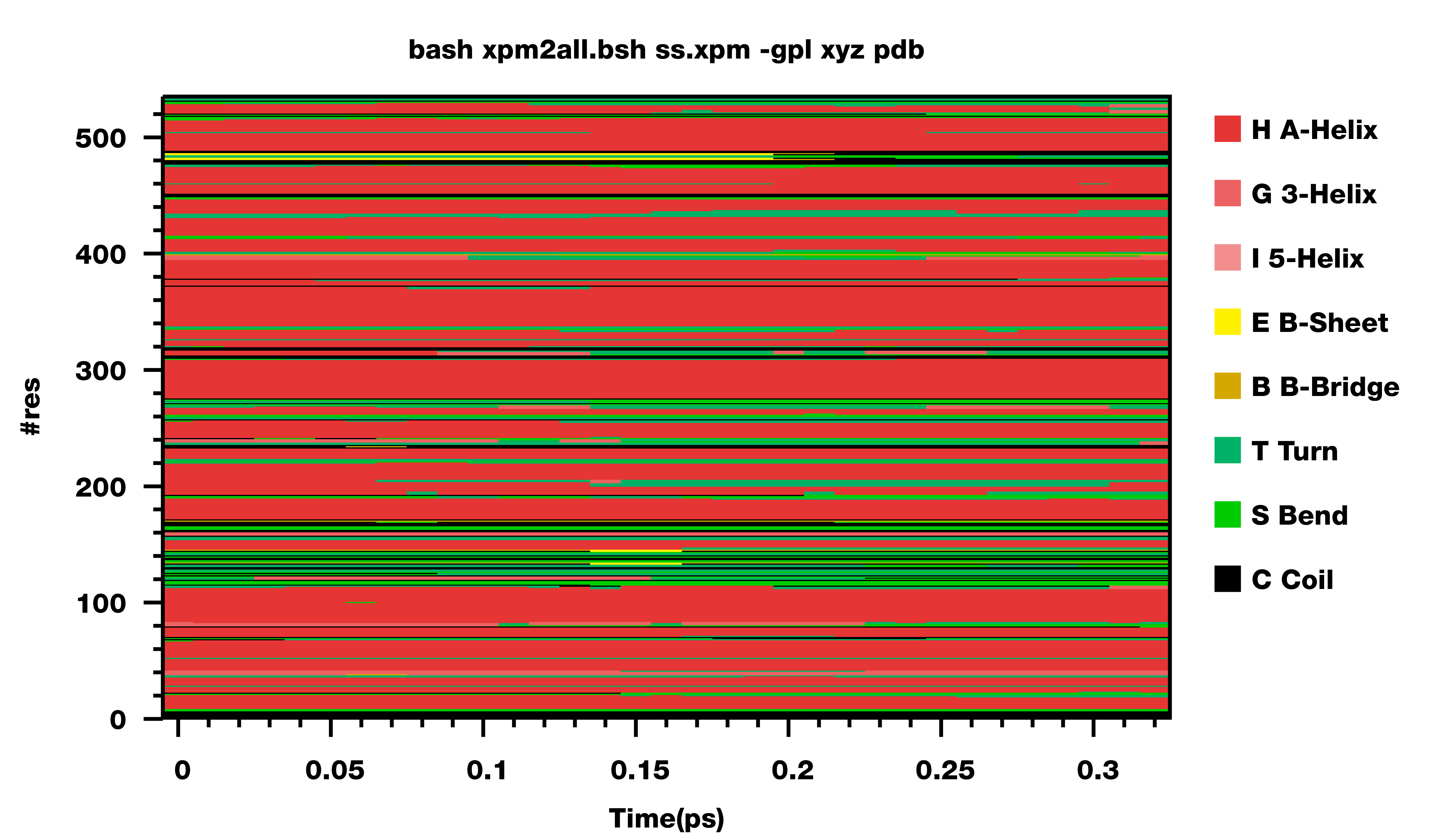
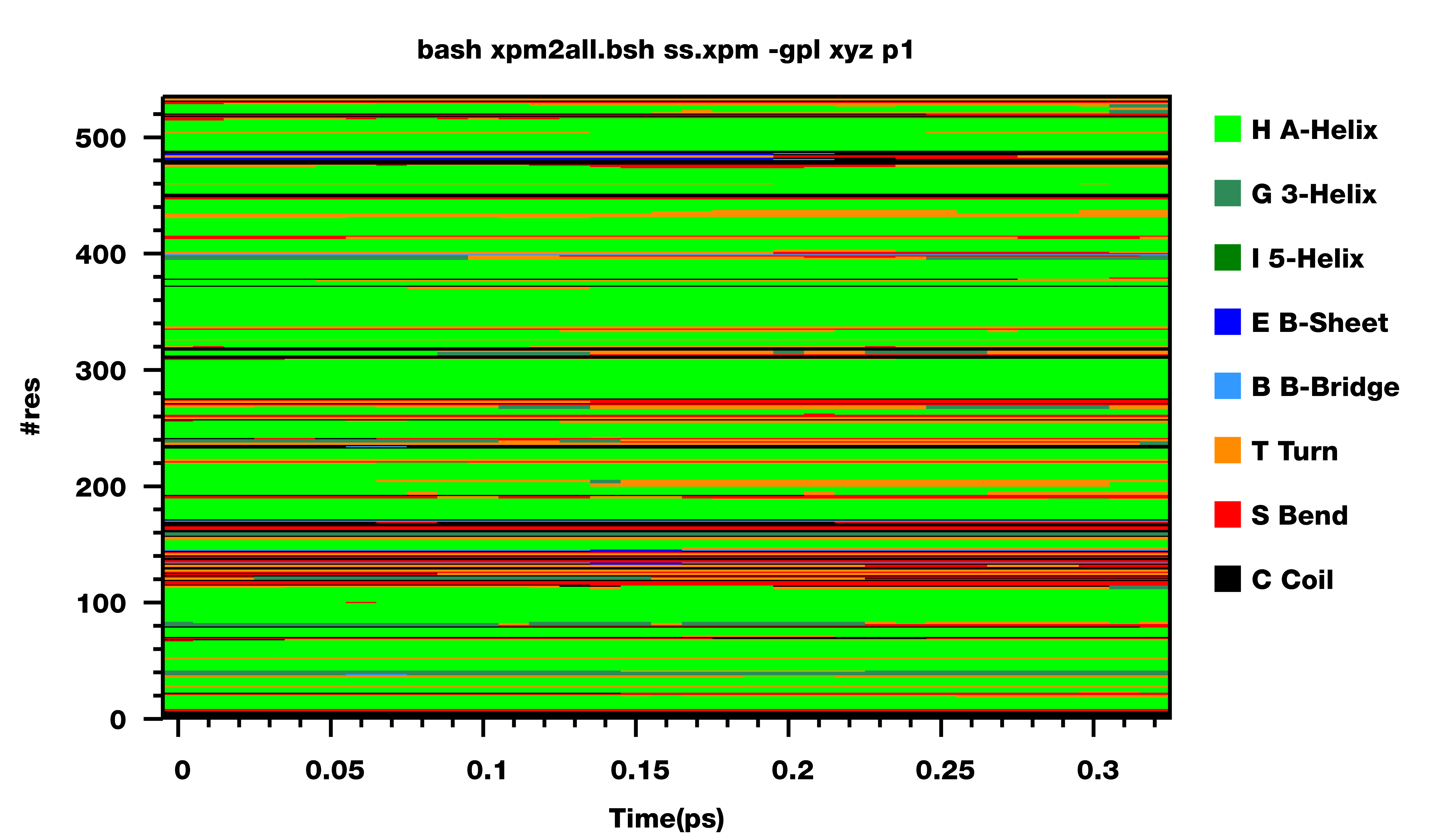
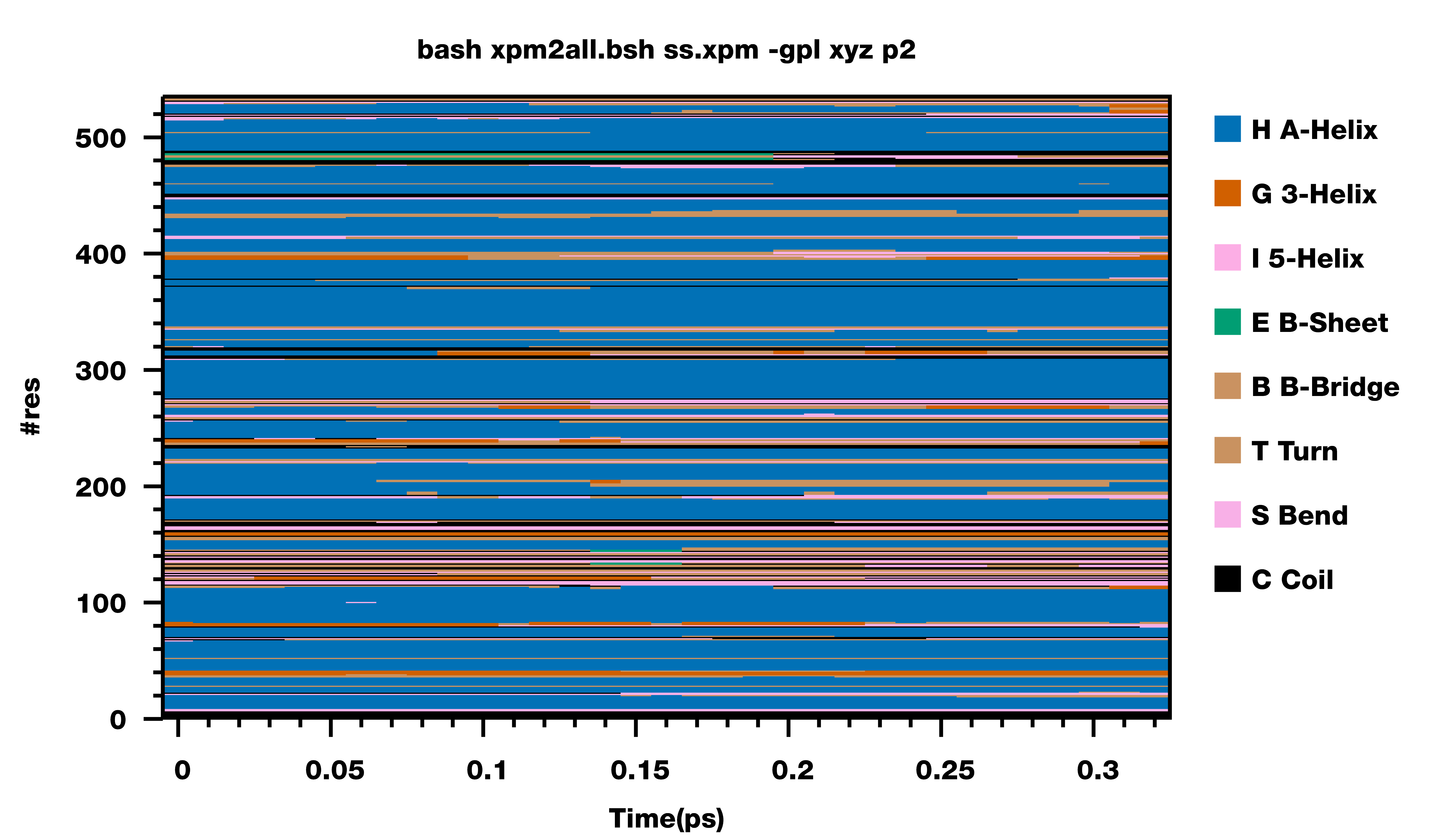
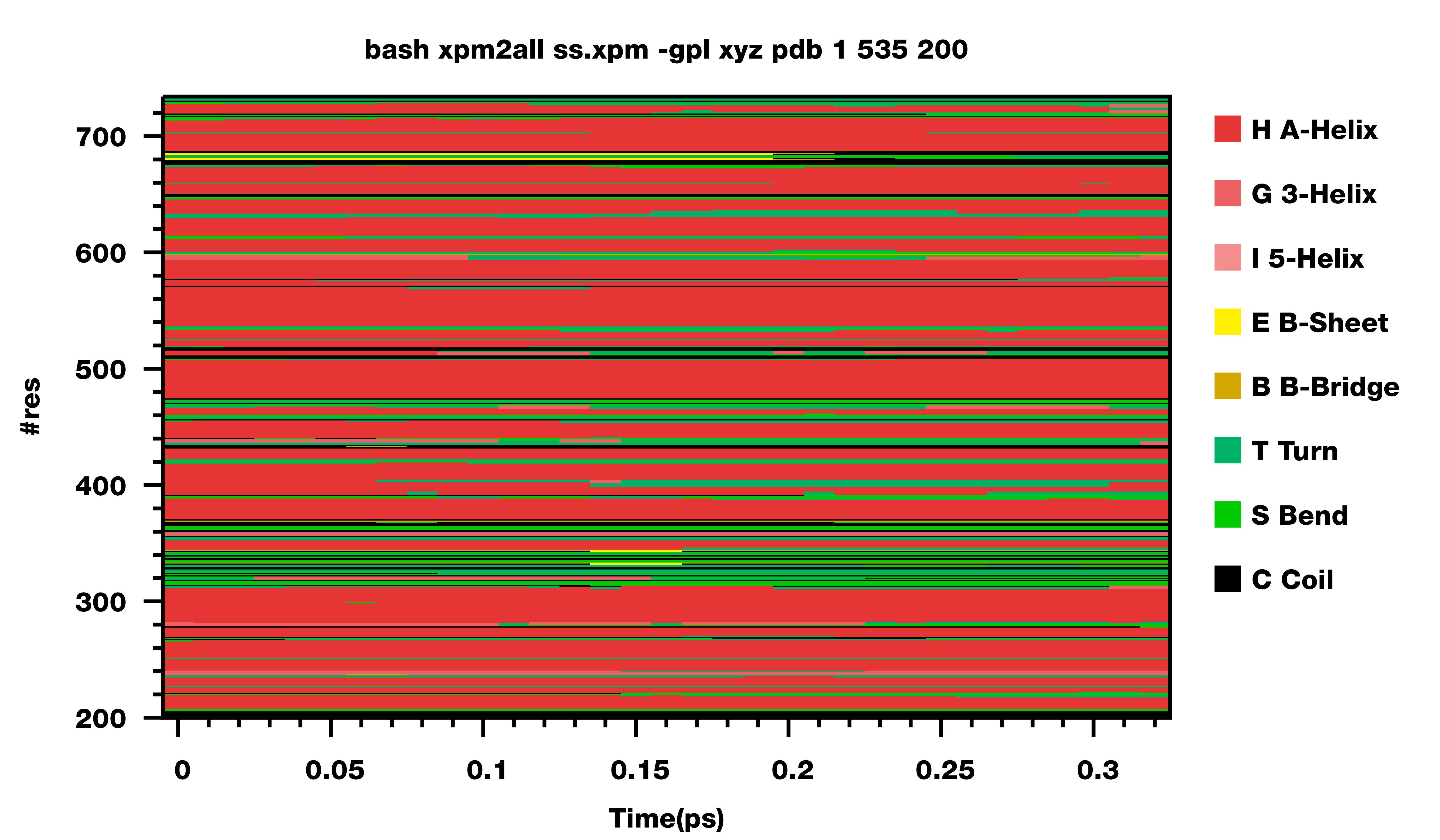
此外, gmx do_dssp输出文件ss.xpm中的残基是你选择要分析的残基, 编号总是从1开始. 如果你分析时选择的并不是所有残基, 那么绘制时的残基编号就与原始蛋白文件中的残基编号不一致了. 为此, 我给脚本增加了另一个选项, 可以指定绘制时残基的编号从什么值开始. 对上面的例子, 如果分析的残基是从200开始的, 而不是从1开始, 那应该如下
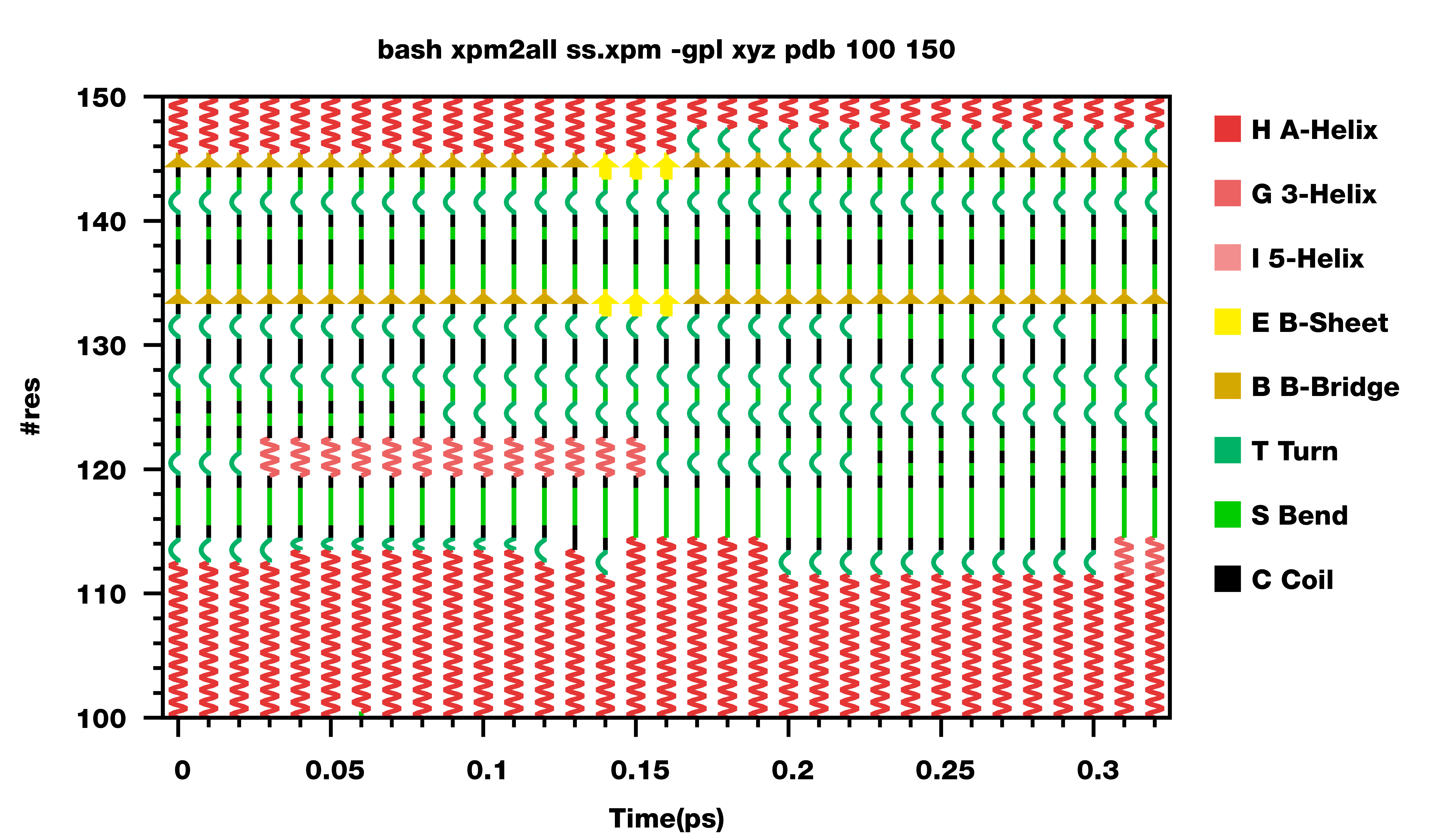
最后, 我还将gnuplot的绘制代码修改了一下, 更方便使用. 当然, fancy的绘制方式还是保留了, 但只用于残基数比较少, 模拟时间比较短的情况.
对于得到的二级结构含量文件ss~count.xvg, 除了使用普通的点线图绘制以外, 还可以使用堆积柱状图绘制. gnuplot支持堆积柱状图, 但其横坐标只能是字符串, 而不支持连续的数值. 这有点不好用. 一个解决方法是对数据稍加处理, 然后使用boxes绘制. 示例如下
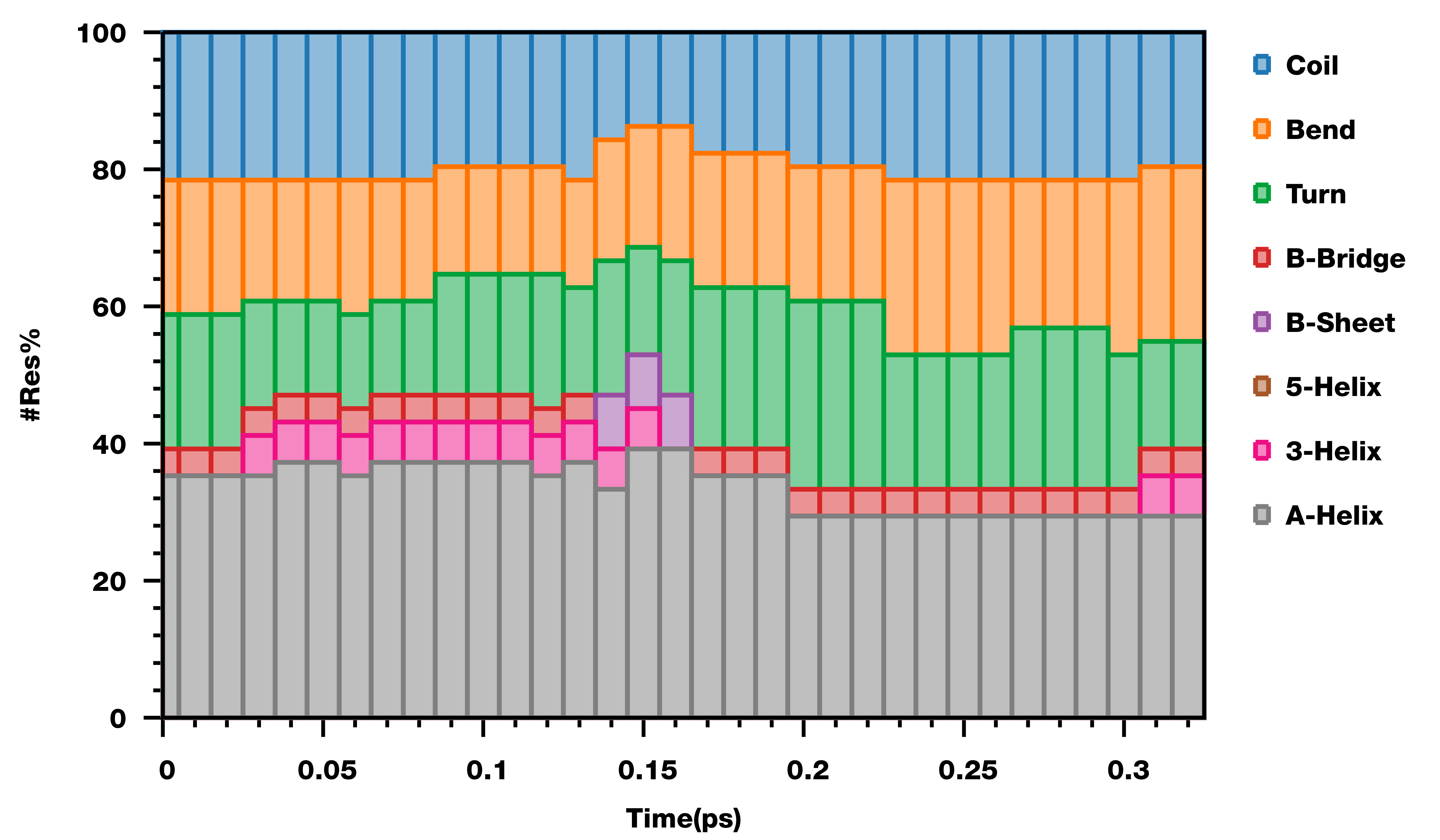
set term pngcairo enhanced truecolor font "HelveticaNeueLT Pro 85 Hv,85" \
fontscale 1 linewidth 20 pointscale 5 size 6000,3500
set tics out nomirror;
set key out reverse Left spacing 2 samplen 1/5
set style fill solid 0.5 border
set xl"Time(ps)"; set yl"#Res%"
plot [0:0.325] [0:100] 'ss~count.xvg' \
u 1:(($2+$3+$4+$5+$6+$7+$8+$9)*100/51) s f w boxes t"Coil ", \
'' u 1:(($2+$3+$4+$5+$6+$7+$8 )*100/51) s f w boxes t"Bend ", \
'' u 1:(($2+$3+$4+$5+$6+$7 )*100/51) s f w boxes t"Turn ", \
'' u 1:(($2+$3+$4+$5+$6 )*100/51) s f w boxes t"B-Bridge", \
'' u 1:(($2+$3+$4+$5 )*100/51) s f w boxes t"B-Sheet ", \
'' u 1:(($2+$3+$4 )*100/51) s f w boxes t"5-Helix ", \
'' u 1:(($2+$3 )*100/51) s f w boxes t"3-Helix ", \
'' u 1:(($2 )*100/51) s f w boxes t"A-Helix "目前就这些了.
