- 2021-06-26 22:37:01
在研究蛋白结构或对蛋白进行分子动力学模拟时, 有时需要对蛋白的二级结构进行评估, 主要是看看拉氏图中的二面角φ, ψ是否在合理范围内. 一个在线的评估工具是PROCHECK, 可以计算给定PDB文件的所有φ, ψ二面角, 然后给出总的评估结果. 这个工具年纪很大了, 所用的评估标准数据也没有更新, 但仍有一定的参考价值, 有些文章中也会用到.
在使用PROCHECK评估蛋白二级结构的时候, 有人提出这样一个问题: 对于来自晶体结构或同源建模的蛋白, 先用PROCHECK进行评估, 跑MD后取平衡的一帧再进行评估, 所得评估结果怎么会变差?
对此, 我的回答是, PROCHECK的评估标准是根据对晶体结构的分析得到的, 所以对蛋白的初始晶体结构, 只要分辨率不是太差, 评估结果一般都比较好. 但对于MD后的某一帧蛋白结构, 即便处于平衡状态, 有些二面角可能波动很大, 再加上所有二面角很难同时处于平衡值, 所以评估结果变差也很有可能. 因此, 真要对MD模拟后的蛋白结构进行评估, 建议使用一段平衡轨迹中二面角的平均值或中位数. 如果发现评估结果仍然不理想, 那很可能是力场的问题. 实际上, 蛋白力场一直在发展, 其中的关注的重要一项就是二面角对实验结果的偏离. 此外, 对于从头建模的结构, 由于初始二面角可能不会接近晶体中的值, 所以MD后评估结果变好的可能性更大.
有鉴于以上, 本文的目的有二: 一是用gnuplot重现PROCHECK评估所给的拉氏图, 一是使用一个小蛋白1crn进行MD模拟, 并对其初始晶体结构, 平衡后某帧结构, 平衡后二面角平均值对应的结构进行评估, 看结果如何.
gnuplot重现PROCHECK的拉氏图
PROCHECK评估结果的拉氏图长得这样
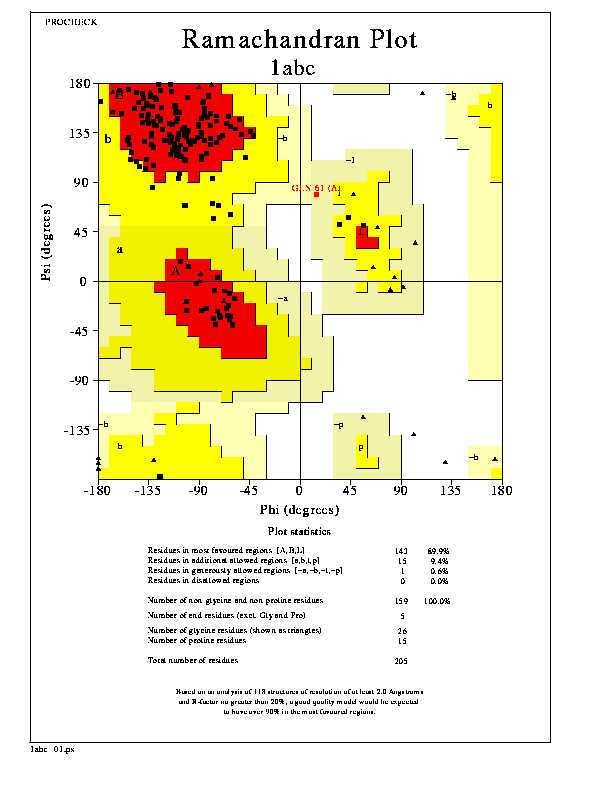
PROCHECK的原始FORTRAN代码中直接用postscript绘制图片, 对研究postscript的人还有参考意义. 不过现在一般没有什么人用这么复杂的方法了. 我就将其用到的数据从源码中抽取出来, 写了几段gnuplot代码绘制类似的图. 为了尽可能地复现, 也添加了边缘线, 标注. 主要目的是练习gnuplot的绘图功能.
gnuplot绘制的图如下
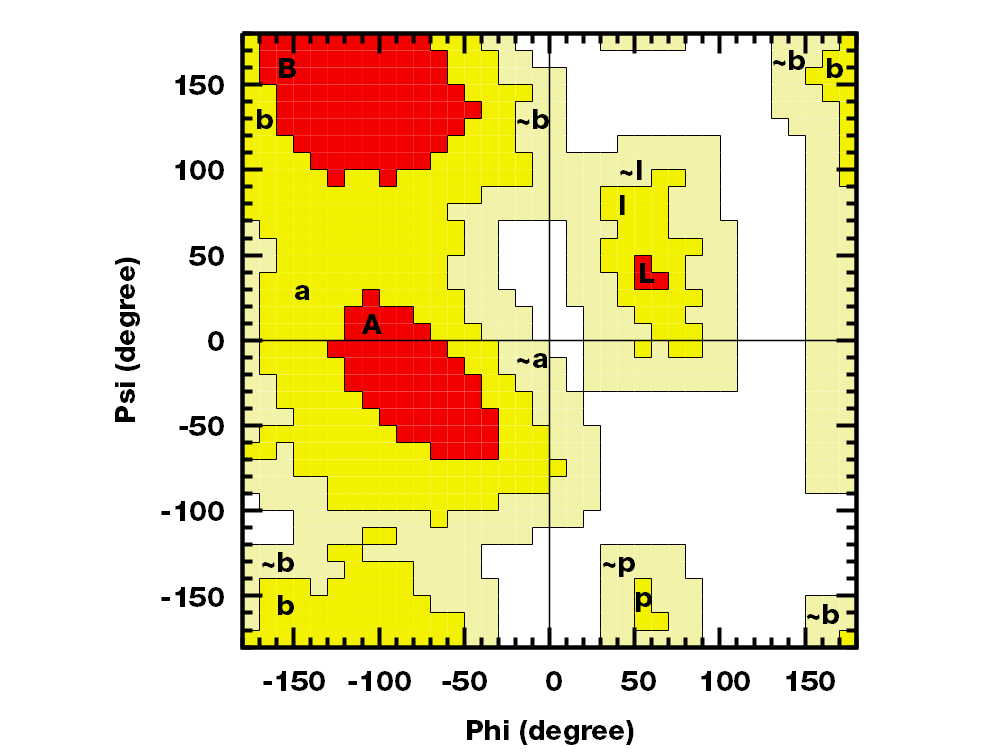
评估蛋白1crn
对小蛋白1crn进行预平衡, 成品NPT模拟, 成品模拟我只进行了1 ns, 看RMSD已经收敛得比较好了.
使用gmx rama计算初始晶体结构, 1 ns结构的拉氏二面角, 借助gnuplot脚本进行评估.
首先是晶体结构的评估结果

1 ns结构的评估结果

接下来计算整段轨迹的二面角. 可以抽取每个残基的二面角看看其变化情况. 我们看看第二个残基THR-2吧.

变化幅度还是有点大的.
最后, 可以借助一个小脚本计算1 ns轨迹的平均二面角, 然后使用平均二面角进行评估.
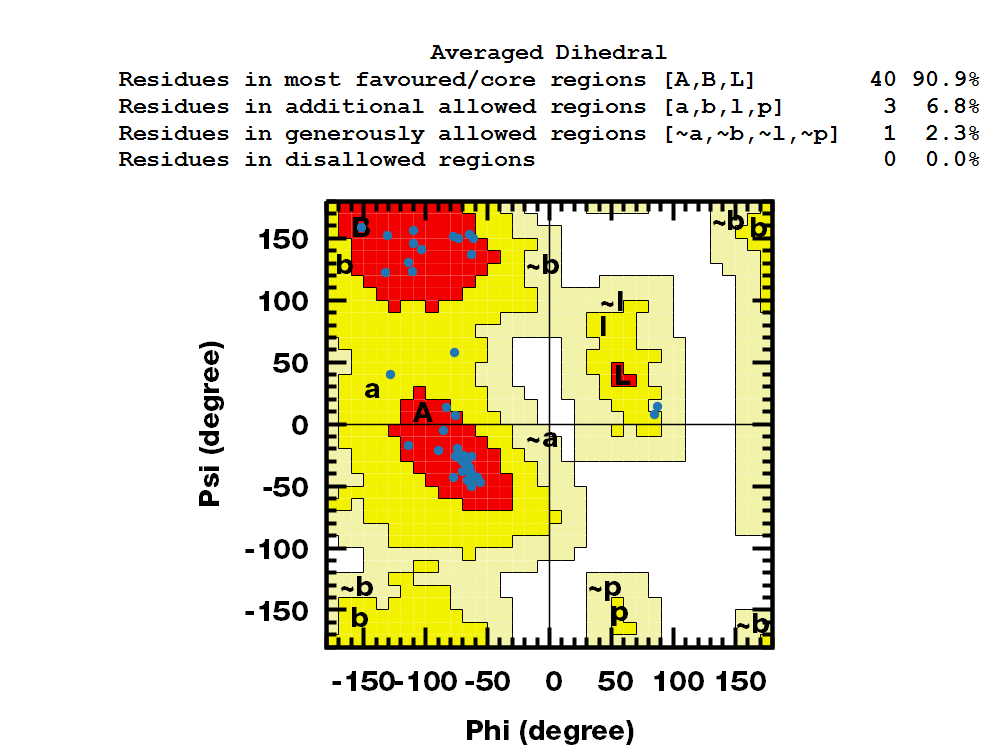
可以看到, 对这个小蛋白来说, MD之后使用某一帧的二面角还是平均二面角, 评估结果相似, 与晶体结构相比都稍有改进, 这可能是因为这个蛋白很小, 且二级结构比较稳定, 也可能和模拟时间过短有关.
先就这样吧.
