- 2022-01-06 16:29:39
- 2022-08-10 21:27:45 感谢 周鼎 测试并提供改进意见
几年前, 我写过几篇GROMACS处理非标准残基的教程, 用的是比较正统的方法, 创建非标准残基的rtp文件, 这样基本上可以一劳永逸, 需要的时候只要pdb2gmx就可以了. 但这种方法还是有点繁琐, 步骤很多, 要注意的地方也很多, 一不留心就可能出错. 其次, 这种方法只适合常规的蛋白修饰, 非标准残基只能与前后两个残基相连, 没法使用这种方法将一个非标准残基的两端连接到任意其他残基上. 再者, 有时候我们只是单纯地有一个非标准残基需要处理, 也没想着还会使用它很多次, 或者将它用到别处, 所做的基本上是个一次性的行为. 在这种情况下, 我们用到非标准残基的时候随手创建一下它的拓扑就好了, 就不劳生成什么rtp了, 那还挺麻烦的. 不借助rtp而直接生成包括非标准残基在内的整个体系的拓扑也是可行的, 虽然步骤可能也还是有些繁琐. 我觉得至少有三种方法可以达到目的: 一种是基于ambertools的建模工具和acpype, 一种是基于片段的方法, 一种是直接修改已有拓扑的方法. 第一种暂且不去说它吧, 第二种比较通用, 可以考虑集成到我的gmxtop工具中, 最后一种稍麻烦点, 需要借助一些脚本来完成, 但比起创建rtp的方法还是简单些, 也可以借以理解拓扑的概念, 熟悉拓扑的修改流程, 所以我就花时间写了几个脚本, 以期能达到目的. 由于时间有限, 并没有将几个脚本综合起来并尽量自动化, 因为那与我期望的目的不合, 脚本也会丧失一些通用性.
基础知识
无论创建拓扑还是修改拓扑, 都要先理解拓扑, 理解非键相互作用, 成键相互作用, 其中重点是成键相互作用, 因为非标准残基麻烦的地方就在于会涉及大量成键项原子编号和参数的处理, 除非是只对标准残基进行非常小的修改, 否则大量成键项的处理必须借助脚本或其他自动工具来减轻工作量, 避免错误.
脚本
这几个脚本暂时只能基于我自己的NotepadX运行. 一共有5个脚本, 其中功能相反的脚本实际上可以合并, 通过选项来指定是删除还是保留, 这里明确分开只是为了简单. 此外, 在这篇教程中我们只会用到其中前3个脚本.
2-1_调整top原子编号.js: 调整拓扑中的原子编号2-2_删除top原子.js: 删除拓扑中某些原子及其涉及的成键项2-3_保留top原子.js: 保留拓扑中某些原子及其涉及的成键项2-4_删除top成键项.js: 删除拓扑中的键及其涉及的键角, 二面角项2-5_添加top成键项.js: 添加键及其涉及的键角, 二面角项
如果删除一个原子, 所有涉及删除原子的成键项也必须删除.
如果删除一条键, 所有涉及此键的键角, 二面角项也需要删除. 所涉及的原子是与两个成键原子二级相邻的所有原子. 添加键的情况类似.
B11 B12
| /
A11 B1--B13
\ /
A12--A1--A--B--B2
/ \
A13 B3示例如上图, 删除或添加A-B键时, 涉及
- 键长:
A-B - 键角:
A-B-B1,A-B-B2,A-B-B3,B-A-A1 - 二面角:
A11-A1-A-B,A12-A1-A-B,A13-A1-A-B,A1-A-B-B1,A1-A-B-B2,A1-A-B-B3,A-B-B1-B11,A-B-B1-B12,A-B-B1-B13
示例
以我常用的小蛋白1crn做示例吧
这个小蛋白的20号残基原本为20GLY, 我将其侧链与一个常见药物分子布洛芬共价相连, 这样就变成了一个非标准残基, 后文就假定其残基名称为20XXX吧.
1. 获得标准蛋白的拓扑: pdb2gmx
先将原pdb文件中的20XXX修改为标准残基, 用于定位, 具体改为哪种都可以, 为简单起见, 最好使用GLY或ALA. 对我们要处理的情况, 由于20XXX是由20GLY修饰而来, 所以使用原来的GLY就好了.
变为标准残基蛋白后先对pdb文件进行清理, 设置好两性残基的质子化状态, 然后使用pdb2gmx补充缺失的氢原子, 最后生成AMBER力场的拓扑.
使用对生成的拓扑进行短时间的模拟, 确认拓扑正常.
; residue 19 PRO rtp PRO q 0.0
273 N 19 PRO N 273 -0.2548 14.01
274 CT 19 PRO CD 274 0.0192 12.01
275 H1 19 PRO HD1 275 0.0391 1.008
276 H1 19 PRO HD2 276 0.0391 1.008
277 CT 19 PRO CG 277 0.0189 12.01
278 HC 19 PRO HG1 278 0.0213 1.008
279 HC 19 PRO HG2 279 0.0213 1.008
280 CT 19 PRO CB 280 -0.007 12.01
281 HC 19 PRO HB1 281 0.0253 1.008
282 HC 19 PRO HB2 282 0.0253 1.008
283 CT 19 PRO CA 283 -0.0266 12.01
284 H1 19 PRO HA 284 0.0641 1.008
285 C 19 PRO C 285 0.5896 12.01
286 O 19 PRO O 286 -0.5748 16 ; qtot 3
; residue 20 GLY rtp GLY q 0.0
287 N 20 GLY N 287 -0.4157 14.01
288 H 20 GLY H 288 0.2719 1.008
289 CT 20 GLY CA 289 -0.0252 12.01
290 H1 20 GLY HA1 290 0.0698 1.008
291 H1 20 GLY HA2 291 0.0698 1.008
292 C 20 GLY C 292 0.5973 12.01
293 O 20 GLY O 293 -0.5679 16 ; qtot 3
; residue 21 THR rtp THR q 0.0
294 N 21 THR N 294 -0.4157 14.01
295 H 21 THR H 295 0.2719 1.008
296 CT 21 THR CA 296 -0.0389 12.01
297 H1 21 THR HA 297 0.1007 1.008
298 CT 21 THR CB 298 0.3654 12.01
299 H1 21 THR HB 299 0.0043 1.008
300 CT 21 THR CG2 300 -0.2438 12.01
301 HC 21 THR HG21 301 0.0642 1.008
302 HC 21 THR HG22 302 0.0642 1.008
303 HC 21 THR HG23 303 0.0642 1.008
304 OH 21 THR OG1 304 -0.6761 16
305 HO 21 THR HG1 305 0.4102 1.008
306 C 21 THR C 306 0.5973 12.01
307 O 21 THR O 307 -0.5679 16 ; qtot 3检查生成的拓扑文件和构型文件, 发现补充完整后蛋白的总原子数为642, 其中19PRO的原子编号从273开始, 20GLY的编号范围为287:293, 21THR从294开始. 记下这些数字, 后面会用到.
2. 删除蛋白拓扑中用于定位非标准残基的标准残基: 2-2_删除top原子.js
由于我们要使用20XXX替换原本的20GLY, 所以先将20GLY的所有原子及其参与的成键项删除. 为此, 选中拓扑中[ atoms ]到[ dihedrals ]的内容, 然后启动脚本2-2_删除top原子.js
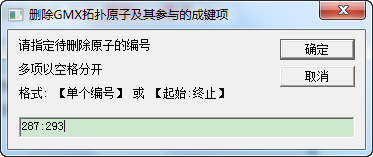
可以直接使用所得结果替换原先选中内容. 如果还不熟悉整个流程, 可以另存为一个文件, 以便与原拓扑对比. 熟悉流程之后, 直接在原文件上修改即可.
与原始拓扑对比一下, 可以发现[ atoms ]中20GLY对应的原子已经删除了.
; residue 19 PRO rtp PRO q 0.0
273 N 19 PRO N 273 -0.2548 14.01
274 CT 19 PRO CD 274 0.0192 12.01
275 H1 19 PRO HD1 275 0.0391 1.008
276 H1 19 PRO HD2 276 0.0391 1.008
277 CT 19 PRO CG 277 0.0189 12.01
278 HC 19 PRO HG1 278 0.0213 1.008
279 HC 19 PRO HG2 279 0.0213 1.008
280 CT 19 PRO CB 280 -0.007 12.01
281 HC 19 PRO HB1 281 0.0253 1.008
282 HC 19 PRO HB2 282 0.0253 1.008
283 CT 19 PRO CA 283 -0.0266 12.01
284 H1 19 PRO HA 284 0.0641 1.008
285 C 19 PRO C 285 0.5896 12.01
286 O 19 PRO O 286 -0.5748 16 ; qtot 3
; residue 20 GLY rtp GLY q 0.0
; residue 21 THR rtp THR q 0.0
294 N 21 THR N 294 -0.4157 14.01
295 H 21 THR H 295 0.2719 1.008
296 CT 21 THR CA 296 -0.0389 12.01
297 H1 21 THR HA 297 0.1007 1.008
298 CT 21 THR CB 298 0.3654 12.01
299 H1 21 THR HB 299 0.0043 1.008
300 CT 21 THR CG2 300 -0.2438 12.01
301 HC 21 THR HG21 301 0.0642 1.008
302 HC 21 THR HG22 302 0.0642 1.008
303 HC 21 THR HG23 303 0.0642 1.008
304 OH 21 THR OG1 304 -0.6761 16
305 HO 21 THR HG1 305 0.4102 1.008
306 C 21 THR C 306 0.5973 12.01
307 O 21 THR O 307 -0.5679 16 ; qtot 3此外, 后面所有涉及20GLY原子的成键项也已经删除.
3. 调整蛋白拓扑的原子编号, 留出非标准残基的位置: 2-1_调整top原子编号.js
20XXX的拓扑最终要合并到蛋白拓扑中, 所以必须先在蛋白拓扑中为其预留下位置, 也就是要调整蛋白拓扑的原子编号, 使其与最终拓扑中的一致.
20XXX共38个原子, 所以蛋白拓扑中要空出38个位置. 因此, 从21THR的第一个原子N294开始, 后续所有原子编号都要进行修改, 294要变为286+38+1=325. 为此, 我们像上一步一样, 选中拓扑中的原子及成键项, 然后启动脚本2-1_调整top原子编号.js
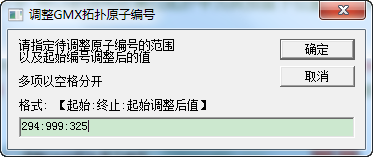
因为我们要修改N294及其后面所有原子的编号, 所以终止编号足够大即可.
将所得结果替换原先选中内容, 就得到了所需的蛋白拓扑.
; residue 19 PRO rtp PRO q 0.0
273 N 19 PRO N 273 -0.2548 14.01
274 CT 19 PRO CD 274 0.0192 12.01
275 H1 19 PRO HD1 275 0.0391 1.008
276 H1 19 PRO HD2 276 0.0391 1.008
277 CT 19 PRO CG 277 0.0189 12.01
278 HC 19 PRO HG1 278 0.0213 1.008
279 HC 19 PRO HG2 279 0.0213 1.008
280 CT 19 PRO CB 280 -0.007 12.01
281 HC 19 PRO HB1 281 0.0253 1.008
282 HC 19 PRO HB2 282 0.0253 1.008
283 CT 19 PRO CA 283 -0.0266 12.01
284 H1 19 PRO HA 284 0.0641 1.008
285 C 19 PRO C 285 0.5896 12.01
286 O 19 PRO O 286 -0.5748 16 ; qtot 3
; residue 20 GLY rtp GLY q 0.0
; residue 21 THR rtp THR q 0.0
325 N 21 THR N 294 -0.4157 14.01
326 H 21 THR H 295 0.2719 1.008
327 CT 21 THR CA 296 -0.0389 12.01
328 H1 21 THR HA 297 0.1007 1.008
329 CT 21 THR CB 298 0.3654 12.01
330 H1 21 THR HB 299 0.0043 1.008
331 CT 21 THR CG2 300 -0.2438 12.01
332 HC 21 THR HG21 301 0.0642 1.008
333 HC 21 THR HG22 302 0.0642 1.008
334 HC 21 THR HG23 303 0.0642 1.008
335 OH 21 THR OG1 304 -0.6761 16
336 HO 21 THR HG1 305 0.4102 1.008
337 C 21 THR C 306 0.5973 12.01
338 O 21 THR O 307 -0.5679 16 ; qtot 3对比检查一下, 确认原子及成键项的编号调整正确.
这样我们就处理好了蛋白部分的拓扑, 等着补充暂时缺失的20XXX拓扑及其成键项.
4. 抽取非标准残基及其两端所连残基, 封端, 获取其GAFF拓扑: ambertools
现在开始处理20XXX. 它处于肽链之中, 前后与其他残基相连, 以常用的NC端顺序而言, 其前端与19PRO的C端相连, 其后端与21THR的N端相连. 我们先将19PRO, 20XXX, 21THR三个残基从PDB中抽取出来
这里之所以不只使用20XXX, 是因为后面将20XXX的拓扑补充到蛋白拓扑中时, 会涉及20XXX与19PRO, 21THR的成键项. 如果只使用20XXX, 有些相应的成键项参数我们要自己补充, 很麻烦, 而使用了前后残基的话, 所有成键参数很容易得到, 后面只要简单复制到蛋白拓扑中就好了.
蛋白我们用了AMBER力场, 非标准残基我们可以使用GAFF力场. 要获得GAFF拓扑首先必须保证分子完整, 也就是要对截取的分子进行封端. 封端的原则是尽量保证其末端原子的化学环境与实际分子中的情况接近. 对于蛋白, 一般是将与C端相连的位置用ACE(-COCH3)封端, 与N端相连位置用NME(-NHCH3)封端.
使用熟悉的分子编辑软件进行封端. 为了后面处理方便, 建议调整一下封端基团的位置, 将ACE放在最前面, NME放在最后面, 也就是按ACE, 19PRO, 20XXX, 21THR, NME的顺序排列原子. 也要注意, 19PRO和21THR自身的原子排列顺序要和前面蛋白中的一致, 否则后面没法保证添加的成键项编号一致.
封端后, 在创建GAFF拓扑前, 一般要使用分子编辑软件进行简单的优化, 使得初始结构尽量合理.
后文我们称这个分子为LIG.
接下来获得LIG分子的GAFF拓扑, 作为测试, 用AM1-BCC电荷就够了. 如果使用RESP电荷, 可以参考我的另一篇教程使用AmberTools+ACPYPE+Gaussian创建小分子GAFF力场的拓扑文件.
可以运行短时间的MD简单测试下所得的GAFF拓扑, 看是否存在问题.
5. 修正非标准残基的电荷
一般情况下, 每个残基的净电荷都是整数, 不带电的残基净电荷是零. 20XXX整体不带电, 所以其38个原子所带电荷之和必须为零, 但由于LIG分子中含有前后残基和封端基团, GAFF只能保证所有原子的净电荷正确, 无法保证20XXX的总电荷为零, 所以必须对20XXX原子的电荷进行修正.
修正的方法很基本, ACE, 19PRO各自的总电荷应该接近零, 21THR, NME各自的总电荷也应该接近零, 所以, 将ACE+19PRO, 21THR+NME从拓扑中删除后, 它们各自的总电荷要想办法分配到20XXX的原子上. 分配的方法也有多种, 既可以直接分配到连接原子上, 也可以平均分配到所有原子上.
查看LIG拓扑的原子部分
[ atoms ]
; nr type resi res atom cgnr charge mass ; qtot bond_type
1 c 1 lig C 1 0.654097 12.01000 ; qtot 0.654
2 o 1 lig O 2 -0.588101 16.00000 ; qtot 0.066
3 c3 1 lig C1 3 -0.175100 12.01000 ; qtot -0.109
4 hc 1 lig H 4 0.072700 1.00800 ; qtot -0.036
5 hc 1 lig H1 5 0.072700 1.00800 ; qtot 0.036
6 hc 1 lig H2 6 0.072700 1.00800 ; ACE qtot 0.109
7 n 1 lig N 7 -0.475800 14.01000 ; qtot -0.367
8 c3 1 lig C2 8 0.078000 12.01000 ; qtot -0.289
9 h1 1 lig H3 9 0.062700 1.00800 ; qtot -0.226
10 h1 1 lig H4 10 0.062700 1.00800 ; qtot -0.163
11 c3 1 lig C3 11 -0.099400 12.01000 ; qtot -0.263
12 hc 1 lig H5 12 0.058200 1.00800 ; qtot -0.205
13 hc 1 lig H6 13 0.058200 1.00800 ; qtot -0.146
14 c3 1 lig C4 14 -0.090400 12.01000 ; qtot -0.237
15 hc 1 lig H7 15 0.067200 1.00800 ; qtot -0.170
16 hc 1 lig H8 16 0.067200 1.00800 ; qtot -0.102
17 c3 1 lig C5 17 0.025700 12.01000 ; qtot -0.077
18 h1 1 lig H9 18 0.088700 1.00800 ; qtot 0.012
19 c 1 lig C6 19 0.656101 12.01000 ; qtot 0.668
20 o 1 lig O1 20 -0.624101 16.00000 ; PRO qtot 0.044
21 n 1 lig N1 21 -0.551901 14.01000 ; qtot -0.508
22 hn 1 lig H10 22 0.328500 1.00800 ; qtot -0.179
23 c3 1 lig C7 23 0.050700 12.01000 ; qtot -0.129
24 h1 1 lig H11 24 0.088700 1.00800 ; qtot -0.040
25 c 1 lig C8 25 0.640101 12.01000 ; qtot 0.600
26 o 1 lig O2 26 -0.639101 16.00000 ; qtot -0.039
27 ca 1 lig C9 27 -0.129000 12.01000 ; qtot -0.168
28 ca 1 lig C10 28 -0.112000 12.01000 ; qtot -0.280
29 ca 1 lig C11 29 -0.095300 12.01000 ; qtot -0.375
30 ca 1 lig C12 30 -0.112000 12.01000 ; qtot -0.487
31 ca 1 lig C13 31 -0.129000 12.01000 ; qtot -0.616
32 ca 1 lig C14 32 -0.069300 12.01000 ; qtot -0.686
33 ha 1 lig H12 33 0.133000 1.00800 ; qtot -0.553
34 ha 1 lig H13 34 0.144000 1.00800 ; qtot -0.409
35 ha 1 lig H14 35 0.144000 1.00800 ; qtot -0.265
36 ha 1 lig H15 36 0.133000 1.00800 ; qtot -0.132
37 c3 1 lig C15 37 -0.056400 12.01000 ; qtot -0.188
38 hc 1 lig H16 38 0.086700 1.00800 ; qtot -0.101
39 c3 1 lig C16 39 -0.091100 12.01000 ; qtot -0.192
40 hc 1 lig H17 40 0.051367 1.00800 ; qtot -0.141
41 hc 1 lig H18 41 0.051367 1.00800 ; qtot -0.090
42 hc 1 lig H19 42 0.051367 1.00800 ; qtot -0.038
43 c 1 lig C17 43 0.641101 12.01000 ; qtot 0.603
44 o 1 lig O3 44 -0.552001 16.00000 ; qtot 0.051
45 oh 1 lig O4 45 -0.606101 16.00000 ; qtot -0.555
46 ho 1 lig H20 46 0.445000 1.00800 ; qtot -0.110
47 c3 1 lig C18 47 -0.041100 12.01000 ; qtot -0.151
48 c3 1 lig C19 48 -0.061700 12.01000 ; qtot -0.213
49 c3 1 lig C20 49 -0.092100 12.01000 ; qtot -0.305
50 hc 1 lig H21 50 0.056700 1.00800 ; qtot -0.249
51 hc 1 lig H22 51 0.039367 1.00800 ; qtot -0.209
52 hc 1 lig H23 52 0.039367 1.00800 ; qtot -0.170
53 hc 1 lig H24 53 0.039367 1.00800 ; qtot -0.130
54 c3 1 lig C21 54 -0.103400 12.01000 ; qtot -0.234
55 hc 1 lig H25 55 0.075200 1.00800 ; qtot -0.159
56 hc 1 lig H26 56 0.075200 1.00800 ; qtot -0.083
57 hc 1 lig H27 57 0.052700 1.00800 ; qtot -0.031
58 hc 1 lig H28 58 0.052700 1.00800 ; XXX qtot 0.022
59 n 1 lig N2 59 -0.535901 14.01000 ; qtot -0.514
60 hn 1 lig H29 60 0.355500 1.00800 ; qtot -0.158
61 c3 1 lig C22 61 0.038700 12.01000 ; qtot -0.120
62 h1 1 lig H30 62 0.087700 1.00800 ; qtot -0.032
63 c3 1 lig C23 63 0.137100 12.01000 ; qtot 0.105
64 h1 1 lig H31 64 0.068700 1.00800 ; qtot 0.174
65 c3 1 lig C24 65 -0.133100 12.01000 ; qtot 0.041
66 hc 1 lig H32 66 0.052367 1.00800 ; qtot 0.093
67 hc 1 lig H33 67 0.052367 1.00800 ; qtot 0.145
68 hc 1 lig H34 68 0.052367 1.00800 ; qtot 0.198
69 oh 1 lig O5 69 -0.609801 16.00000 ; qtot -0.412
70 ho 1 lig H35 70 0.410000 1.00800 ; qtot -0.002
71 c 1 lig C25 71 0.645101 12.01000 ; qtot 0.643
72 o 1 lig O6 72 -0.623101 16.00000 ; THR qtot 0.020
73 n 1 lig N3 73 -0.565901 14.01000 ; qtot -0.546
74 c3 1 lig C26 74 0.078300 12.01000 ; qtot -0.468
75 hn 1 lig H36 75 0.340500 1.00800 ; qtot -0.127
76 h1 1 lig H37 76 0.042367 1.00800 ; qtot -0.085
77 h1 1 lig H38 77 0.042367 1.00800 ; qtot -0.042
78 h1 1 lig H39 78 0.042367 1.00800 ; NME qtot -0.000可以看到, 前20个原子为ACE+19PRO, 最后20个原子为21THR+NME. 我们分别将这两组原子的总电荷加到与其相连的原子上. 具体而言, ACE+19PRO总电荷为0.043996, 将其加到N21上, 则N21电荷应为-0.551901+0.043996=-0.507905, 21THR+NME总电荷为-0.022001, 将其加到C25上, 则C25电荷应为0.640101-0.022001=0.6181. 我们可以看到, 这两组原子的总电荷都很接近零, 删除它们对20XXX电荷的影响比较小, 说明我们采用的方法比较合理.
修正电荷后, 我们再验证一下20XXX的总电荷是否为零. 由于数值计算的精度, 舍入, 截断等原因, 有时总电荷并不会恰好为零, 而是有微小的偏差. 对于我们的数据, 修正后20XXX的总电荷为-5e-5, 非常小, 但不是零. 所以, 我们再微调一下. 微调时可以任意选择一个原子的电荷, 给它增加等量的相反电荷, 也可以试着删除所有电荷的最后一位数字, 因为很多时候是最后一位数字导致的偏差. 至于具体选择哪个原子, 采用哪种方式, 没有定例, 怎么方便怎么来吧.
查看我们所得的数字, 删除最后一位数字并不方便, 删除N21电荷的最后一位数字5, 或将C25电荷最后增加05最方便. 这样调整后, 20XXX的总电荷为-1.5e-16, 足够接近零了.
[ atoms ]
; nr type resi res atom cgnr charge mass ; qtot bond_type
1 c 1 lig C 1 0.654097 12.01000 ; qtot 0.654
2 o 1 lig O 2 -0.588101 16.00000 ; qtot 0.066
3 c3 1 lig C1 3 -0.175100 12.01000 ; qtot -0.109
4 hc 1 lig H 4 0.072700 1.00800 ; qtot -0.036
5 hc 1 lig H1 5 0.072700 1.00800 ; qtot 0.036
6 hc 1 lig H2 6 0.072700 1.00800 ; ACE qtot 0.109
7 n 1 lig N 7 -0.475800 14.01000 ; qtot -0.367
8 c3 1 lig C2 8 0.078000 12.01000 ; qtot -0.289
9 h1 1 lig H3 9 0.062700 1.00800 ; qtot -0.226
10 h1 1 lig H4 10 0.062700 1.00800 ; qtot -0.163
11 c3 1 lig C3 11 -0.099400 12.01000 ; qtot -0.263
12 hc 1 lig H5 12 0.058200 1.00800 ; qtot -0.205
13 hc 1 lig H6 13 0.058200 1.00800 ; qtot -0.146
14 c3 1 lig C4 14 -0.090400 12.01000 ; qtot -0.237
15 hc 1 lig H7 15 0.067200 1.00800 ; qtot -0.170
16 hc 1 lig H8 16 0.067200 1.00800 ; qtot -0.102
17 c3 1 lig C5 17 0.025700 12.01000 ; qtot -0.077
18 h1 1 lig H9 18 0.088700 1.00800 ; qtot 0.012
19 c 1 lig C6 19 0.656101 12.01000 ; qtot 0.668
20 o 1 lig O1 20 -0.624101 16.00000 ; PRO qtot 0.044
21 n 1 lig N1 21 -0.50790 14.01000 ; 删除末位的5
22 hn 1 lig H10 22 0.328500 1.00800
23 c3 1 lig C7 23 0.050700 12.01000
24 h1 1 lig H11 24 0.088700 1.00800
25 c 1 lig C8 25 0.6181 12.01000
26 o 1 lig O2 26 -0.639101 16.00000
27 ca 1 lig C9 27 -0.129000 12.01000
28 ca 1 lig C10 28 -0.112000 12.01000
29 ca 1 lig C11 29 -0.095300 12.01000
30 ca 1 lig C12 30 -0.112000 12.01000
31 ca 1 lig C13 31 -0.129000 12.01000
32 ca 1 lig C14 32 -0.069300 12.01000
33 ha 1 lig H12 33 0.133000 1.00800
34 ha 1 lig H13 34 0.144000 1.00800
35 ha 1 lig H14 35 0.144000 1.00800
36 ha 1 lig H15 36 0.133000 1.00800
37 c3 1 lig C15 37 -0.056400 12.01000
38 hc 1 lig H16 38 0.086700 1.00800
39 c3 1 lig C16 39 -0.091100 12.01000
40 hc 1 lig H17 40 0.051367 1.00800
41 hc 1 lig H18 41 0.051367 1.00800
42 hc 1 lig H19 42 0.051367 1.00800
43 c 1 lig C17 43 0.641101 12.01000
44 o 1 lig O3 44 -0.552001 16.00000
45 oh 1 lig O4 45 -0.606101 16.00000
46 ho 1 lig H20 46 0.445000 1.00800
47 c3 1 lig C18 47 -0.041100 12.01000
48 c3 1 lig C19 48 -0.061700 12.01000
49 c3 1 lig C20 49 -0.092100 12.01000
50 hc 1 lig H21 50 0.056700 1.00800
51 hc 1 lig H22 51 0.039367 1.00800
52 hc 1 lig H23 52 0.039367 1.00800
53 hc 1 lig H24 53 0.039367 1.00800
54 c3 1 lig C21 54 -0.103400 12.01000
55 hc 1 lig H25 55 0.075200 1.00800
56 hc 1 lig H26 56 0.075200 1.00800
57 hc 1 lig H27 57 0.052700 1.00800
58 hc 1 lig H28 58 0.052700 1.00800
59 n 1 lig N2 59 -0.535901 14.01000 ; qtot -0.514
60 hn 1 lig H29 60 0.355500 1.00800 ; qtot -0.158
61 c3 1 lig C22 61 0.038700 12.01000 ; qtot -0.120
62 h1 1 lig H30 62 0.087700 1.00800 ; qtot -0.032
63 c3 1 lig C23 63 0.137100 12.01000 ; qtot 0.105
64 h1 1 lig H31 64 0.068700 1.00800 ; qtot 0.174
65 c3 1 lig C24 65 -0.133100 12.01000 ; qtot 0.041
66 hc 1 lig H32 66 0.052367 1.00800 ; qtot 0.093
67 hc 1 lig H33 67 0.052367 1.00800 ; qtot 0.145
68 hc 1 lig H34 68 0.052367 1.00800 ; qtot 0.198
69 oh 1 lig O5 69 -0.609801 16.00000 ; qtot -0.412
70 ho 1 lig H35 70 0.410000 1.00800 ; qtot -0.002
71 c 1 lig C25 71 0.645101 12.01000 ; qtot 0.643
72 o 1 lig O6 72 -0.623101 16.00000 ; THR qtot 0.020
73 n 1 lig N3 73 -0.565901 14.01000 ; qtot -0.546
74 c3 1 lig C26 74 0.078300 12.01000 ; qtot -0.468
75 hn 1 lig H36 75 0.340500 1.00800 ; qtot -0.127
76 h1 1 lig H37 76 0.042367 1.00800 ; qtot -0.085
77 h1 1 lig H38 77 0.042367 1.00800 ; qtot -0.042
78 h1 1 lig H39 78 0.042367 1.00800 ; NME qtot -0.0006. 调整编号, 使其与蛋白拓扑中的一致: 2-1_调整top原子编号.js
20XXX最终是要合并到蛋白拓扑中的, 而且我们前面已经在蛋白拓扑中为它预留了位置, 所以其原子编号必须与最终蛋白拓扑中的一致. 此外, 也需要将前后相连残基19PRO, 21THR的原子编号调整至与蛋白拓扑中的一致, 方便后面添加成键项.
回顾前面的数字, 蛋白拓扑中19PRO的原子编号从273开始, 而LIG中的则从7开始, 所以我们再次使用脚本2-1_调整top原子编号.js, 设定7:999:273, 这样就得到了调整编号后的LIG拓扑.
[ atoms ]
; nr type resi res atom cgnr charge mass ; qtot bond_type
1 c 1 lig C 1 0.654097 12.01000 ; qtot 0.654
2 o 1 lig O 2 -0.588101 16.00000 ; qtot 0.066
3 c3 1 lig C1 3 -0.175100 12.01000 ; qtot -0.109
4 hc 1 lig H 4 0.072700 1.00800 ; qtot -0.036
5 hc 1 lig H1 5 0.072700 1.00800 ; qtot 0.036
6 hc 1 lig H2 6 0.072700 1.00800 ; ACE qtot 0.109
273 n 1 lig N 7 -0.475800 14.01000 ; qtot -0.367
274 c3 1 lig C2 8 0.078000 12.01000 ; qtot -0.289
275 h1 1 lig H3 9 0.062700 1.00800 ; qtot -0.226
276 h1 1 lig H4 10 0.062700 1.00800 ; qtot -0.163
277 c3 1 lig C3 11 -0.099400 12.01000 ; qtot -0.263
278 hc 1 lig H5 12 0.058200 1.00800 ; qtot -0.205
279 hc 1 lig H6 13 0.058200 1.00800 ; qtot -0.146
280 c3 1 lig C4 14 -0.090400 12.01000 ; qtot -0.237
281 hc 1 lig H7 15 0.067200 1.00800 ; qtot -0.170
282 hc 1 lig H8 16 0.067200 1.00800 ; qtot -0.102
283 c3 1 lig C5 17 0.025700 12.01000 ; qtot -0.077
284 h1 1 lig H9 18 0.088700 1.00800 ; qtot 0.012
285 c 1 lig C6 19 0.656101 12.01000 ; qtot 0.668
286 o 1 lig O1 20 -0.624101 16.00000 ; PRO qtot 0.044
287 n 1 lig N1 21 -0.50790 14.01000
288 hn 1 lig H10 22 0.328500 1.00800
289 c3 1 lig C7 23 0.050700 12.01000
290 h1 1 lig H11 24 0.088700 1.00800
291 c 1 lig C8 25 0.6181 12.01000
292 o 1 lig O2 26 -0.639101 16.00000
293 ca 1 lig C9 27 -0.129000 12.01000
294 ca 1 lig C10 28 -0.112000 12.01000
295 ca 1 lig C11 29 -0.095300 12.01000
296 ca 1 lig C12 30 -0.112000 12.01000
297 ca 1 lig C13 31 -0.129000 12.01000
298 ca 1 lig C14 32 -0.069300 12.01000
299 ha 1 lig H12 33 0.133000 1.00800
300 ha 1 lig H13 34 0.144000 1.00800
301 ha 1 lig H14 35 0.144000 1.00800
302 ha 1 lig H15 36 0.133000 1.00800
303 c3 1 lig C15 37 -0.056400 12.01000
304 hc 1 lig H16 38 0.086700 1.00800
305 c3 1 lig C16 39 -0.091100 12.01000
306 hc 1 lig H17 40 0.051367 1.00800
307 hc 1 lig H18 41 0.051367 1.00800
308 hc 1 lig H19 42 0.051367 1.00800
309 c 1 lig C17 43 0.641101 12.01000
310 o 1 lig O3 44 -0.552001 16.00000
311 oh 1 lig O4 45 -0.606101 16.00000
312 ho 1 lig H20 46 0.445000 1.00800
313 c3 1 lig C18 47 -0.041100 12.01000
314 c3 1 lig C19 48 -0.061700 12.01000
315 c3 1 lig C20 49 -0.092100 12.01000
316 hc 1 lig H21 50 0.056700 1.00800
317 hc 1 lig H22 51 0.039367 1.00800
318 hc 1 lig H23 52 0.039367 1.00800
319 hc 1 lig H24 53 0.039367 1.00800
320 c3 1 lig C21 54 -0.103400 12.01000
321 hc 1 lig H25 55 0.075200 1.00800
322 hc 1 lig H26 56 0.075200 1.00800
323 hc 1 lig H27 57 0.052700 1.00800
324 hc 1 lig H28 58 0.052700 1.00800
325 n 1 lig N2 59 -0.535901 14.01000 ; qtot -0.514
326 hn 1 lig H29 60 0.355500 1.00800 ; qtot -0.158
327 c3 1 lig C22 61 0.038700 12.01000 ; qtot -0.120
328 h1 1 lig H30 62 0.087700 1.00800 ; qtot -0.032
329 c3 1 lig C23 63 0.137100 12.01000 ; qtot 0.105
330 h1 1 lig H31 64 0.068700 1.00800 ; qtot 0.174
331 c3 1 lig C24 65 -0.133100 12.01000 ; qtot 0.041
332 hc 1 lig H32 66 0.052367 1.00800 ; qtot 0.093
333 hc 1 lig H33 67 0.052367 1.00800 ; qtot 0.145
334 hc 1 lig H34 68 0.052367 1.00800 ; qtot 0.198
335 oh 1 lig O5 69 -0.609801 16.00000 ; qtot -0.412
336 ho 1 lig H35 70 0.410000 1.00800 ; qtot -0.002
337 c 1 lig C25 71 0.645101 12.01000 ; qtot 0.643
338 o 1 lig O6 72 -0.623101 16.00000 ; THR qtot 0.020
339 n 1 lig N3 73 -0.565901 14.01000 ; qtot -0.546
340 c3 1 lig C26 74 0.078300 12.01000 ; qtot -0.468
341 hn 1 lig H36 75 0.340500 1.00800 ; qtot -0.127
342 h1 1 lig H37 76 0.042367 1.00800 ; qtot -0.085
343 h1 1 lig H38 77 0.042367 1.00800 ; qtot -0.042
344 h1 1 lig H39 78 0.042367 1.00800 ; NME qtot -0.0007. 抽取非标准残基的原子及其参与的成键项: 2-3_保留top原子.js
将20XXX合并到蛋白拓扑中时, 需要其自身原子, 自身原子间的成键项, 自身原子与19PRO, 21THR的成键项, 由于这些成键项在LIG的拓扑中都是存在的, 所以我们可以直接抽取出来使用. 这也是我们前面创建LIG拓扑时使用19PRO, 21THR的原因.
使用步骤6中调整好编号的拓扑, 启动脚本2-3_保留top原子.js, 保留287:324
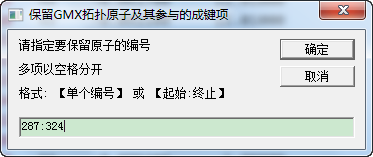
这样就得到了所需的原子及其成键项.
注意: 这一步骤在先前的教程中是通过两步完成的: 先添加需要的成键项获得20XXX与前后残基的成键项, 再删除不属于20XXX的原子获得其自身原子及其成键项. 这种方法可行, 但稍麻烦些.
8. 合并所有非标准残基拓扑到蛋白拓扑
将步骤7所得拓扑[ atoms ]部分复制粘贴到蛋白拓扑的对应位置, 完成最终蛋白拓扑的[ atoms ]部分.
将步骤7所得的成键项, 复制粘贴到蛋白拓扑的相应位置, 完成最终蛋白拓扑的成键部分.
9. 测试所得拓扑
使用得到的拓扑进行短时间的模拟, 确认不存在问题.
由于蛋白中添加了非标准残基, 所以进行位置限制动力学时, 非标准残基原子也要限制. 此外, 设置温度耦合组时要注意将非标准残基包含在蛋白组中.
几点说明
-
此方法看似步骤很多, 是因为我写得详细, 实际操作并不复杂, 难度低于创建rtp的方法. 主要难点在于数数, 数对原子编号.
-
此方法比生成rtp的方法更通用, 几乎可以将任何分子连接到蛋白上.
-
将非标准残基拓扑与蛋白拓扑进行组合时, 可以简单地将非标准残基追加到最后, 但这样做残基编号, 尤其是与非标准残基前后相连的残基编号与GAFF拓扑中的区别较大, 后面获得成键项时会不方便, 所以还是尽量放在原来的对应位置吧.
-
删除非标准残基的蛋白拓扑, 删除封端基团的GAFF拓扑, 对原子编号进行调整, 保证连续后都可以进行简单的模拟, 以验证拓扑是否存在问题.
-
你可以下载添加了布洛芬的
1crn, 自己试着处理 -
你还可以下载另一个我瞎改后的
1crn, 里面使用长的碳链将两个距离很远的残基连起来了. 这种情况也可以处理, 虽然稍微麻烦点.
就这些吧. 完了.
