- 2019-08-05 17:50:22
在文献Tristan Bereau, Kurt Kremer; Automated Parametrization of the Coarse-Grained Martini Force Field for Small Organic Molecules; J. Chem. Theory Comput. 11(6):2783-2791, 2015; 10.1021/acs.jctc.5b00056中提出了自动将小分子划分为Martini珠子的方法, 作者还提供了一个python脚本. 这里简单介绍下它的使用方法.
安装
- 首先从github下载
auto-martini程序包放到合适目录 - 下载Windows版的anaconda2. 注意, 这里要选择python2.7版本的, 因为
auto-martini是用python2.7写的, 不兼容python3之后的语法 - 安装完anaconda之后, 以管理员身份运行anaconda, 进入
auto-martini包所在位置 - 安装
auto-martini所依赖的四个程序包
pip install numpy
pip install beautifulsoup
pip install requests
conda create -c rdkit -n my-rdkit-env rdkit安装完之后, 就可以使用了.
使用方法
命令
python auto-martini [-h] (--sdf SDF | --smi SMI) --mol MOLNAME [--xyz XYZ] [--gro GRO] [--verbose] [--fpred]说明
-
--sdf和--smi: 输入文件, 指定其中一个就可以. 可以使用openbabel将pdb或其他格式的文件转化为sdf或者smi文件. -
--mol: 必须选项, 输出文件中残基的名称 -
--xyz,--gro: 可选的输出文件 -
--verbose,--fpred: 无法找到符合的参数时, 使用按原子或按片段判别珠子的方法, 准确度较差
示例
使用Gaussview画出想要的小分子
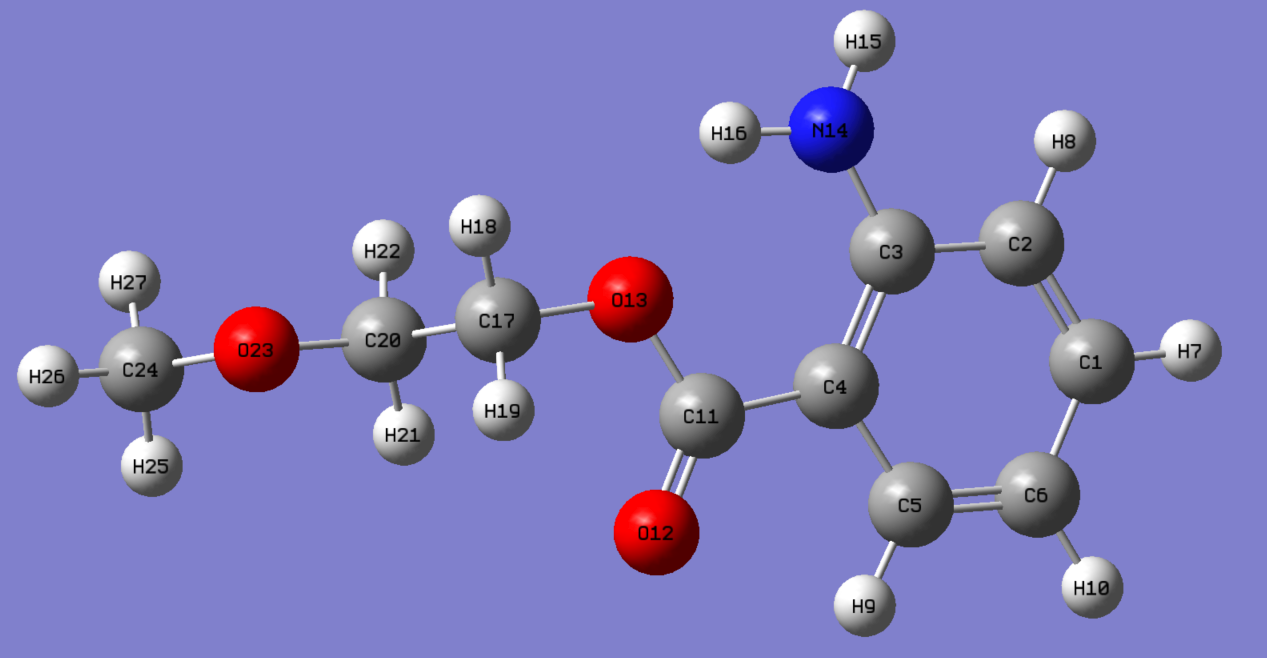
可以直接保存为sdf文件, 但好像Gaussview生成的sdf文件在auto-martini中使用会出错(未详细考察), 所以我们保存为pdb文件, 再用openbabel转换为sdf文件.
将该文件放到auto-martini目录下. 执行
python auto_martini --sdf TEG.sdf --mol TEG --gro TEG_CG.gro会得到如下结果
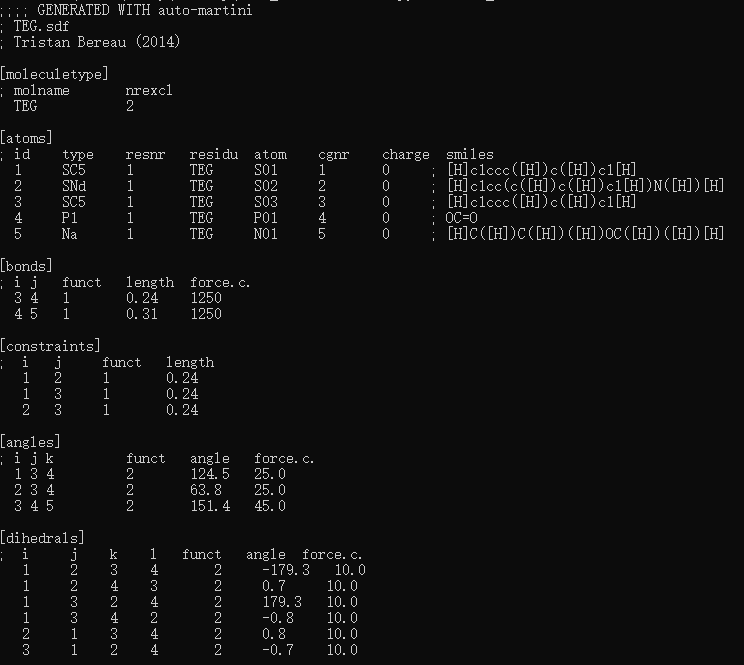
同时生成gro文件

注意事项
- 如果提示
smi2alogps这一步出错, 那你得先检查一下 http://vcclab.org/web/alogps 网站是否能够打开, 因为auto-martini会将你的分子上传到alogps网站上, 生成所需的logP等信息
