- 2021-03-16 22:43:43
我的gmx_mmpbsa脚本发布时间也不短了, 有不少人用过, 也有些人在文章中引用. 但凡一件事物, 用的人多了, 总会暴露出一些问题与缺陷, 于代码而言, 尤其如此. 查看总结网上涉及gmx_mmpbsa的留言与问题后, 我觉得有必要更新一下这个脚本, 其中最主要的一点就是, 试着解决所得结合能数值不合理的问题.
在计算两个组分, 如蛋白和配体的结合能时, 如果其中一个或两个组分带有净电荷, 气相MM部分的静电相互作用MM(COU)往往会很大, 从而导致总的结合能绝对数值过大或为正值. 这并不是什么计算错误, 而是由MMPBSA这个方法的近似导致的. 虽然计算MM(COU)时可以引入溶质的介电常数对此进行校正, 但具体使用什么数值, 文献上并没有定论. 一些讨论可以参考文献[^1].
最近有篇论文[^2]使用gmx_mmpbsa研究了新冠病毒与ACE2, 两种抗体的结合能. 由于涉及到的蛋白都带有很大的净电荷, 直接使用原始MMPBSA方法自然会得到非常大的结合能, 远远大于实验值, 明显与实验结果不符, 而且连相对强弱顺序也与实验结果不一致. 为此, 文章的作者建议使用德拜-休克尔屏蔽方法计算MM(COU): 计算MM(COU)时使用考虑离子强度, 使用德拜特征长度对静电作用进行指数衰减. 这样处理后, 所得MM(COU)贡献就变小了, 再加上考虑熵的贡献, 最终所得的结合能总算与实验值比较一致了, 相对强弱顺序也能对得上了.
我不是专门研究自由能计算的, 所以也不太关注这些, 但鉴于他们是基于自行修改的gmx_mmpbsa脚本进行计算的, 所以我觉得还是将他们的方法集成到我的脚本中去, 弄成类似官方支持吧, 这样如果有人需要使用这种方法, 能更方便一些.
代码
见https://jerkwin.github.io/gmxtools/.
几点说明:
- 这种屏蔽方法实现起来并不复杂, 但需要指出论文[^2]中所给德拜长度公式存在笔误, 根号应该扩展到整个表达式. 此外, 公式中的相对介电常数应该用水的值.
- 脚本将PB计算的默认方法由线性lpbe方法改为非线性npbe方法. 根据一些资料的说法, 对于净电荷很大的体系, lpbe方法误差过大. 根据维基, 改为npbe后, 所得PB相互作用能会变小.
- 再次强调, 网格参数
df对PB结果的影响非常显著, 默认值0.5可能并未达到收敛的结果. 如果要仔细对待结果, 那么请牢记这一点.
简单测试
1EBZ: 屏蔽效应并不显著
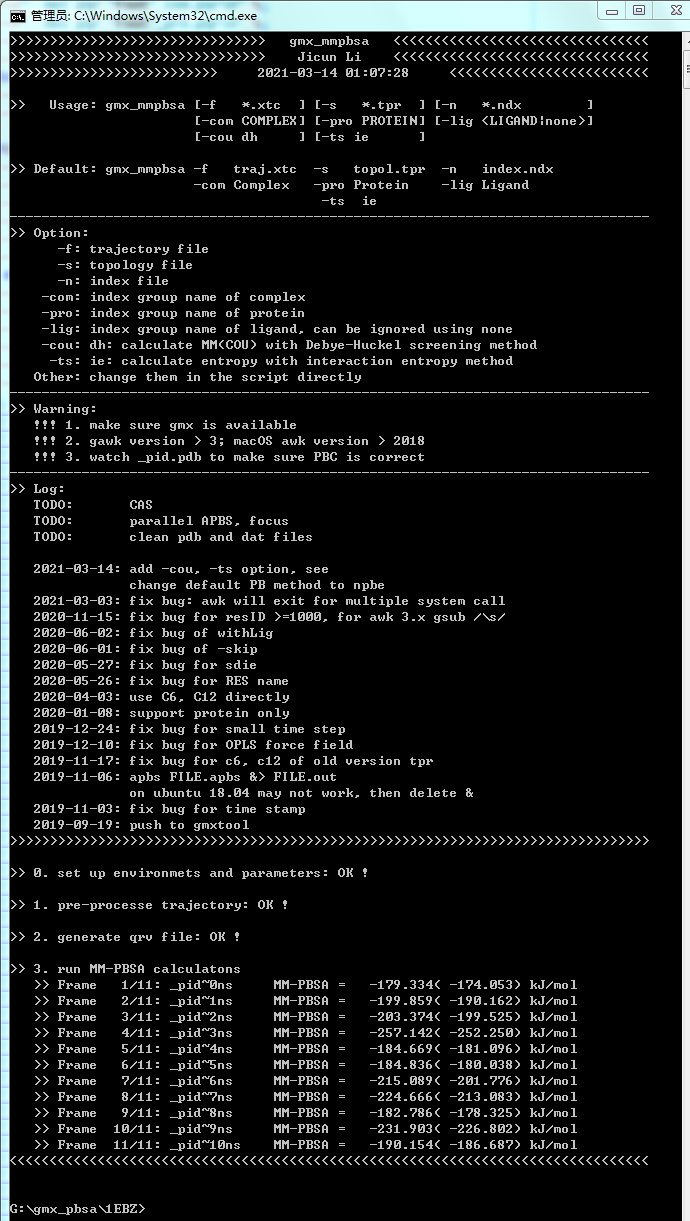
| #Frame | Binding( with DH ) | MM ( with DH ) | PB | SA | COU ( with DH ) | VDW | |
|---|---|---|---|---|---|---|---|
| 0ns | -179.334( -174.053) | -468.203( -462.922) | 323.523 | -34.654 | -147.070( -141.788) | -321.134 | |
| 1ns | -199.859( -190.162) | -488.041( -478.344) | 322.631 | -34.449 | -176.307( -166.610) | -311.734 | |
| 2ns | -203.374( -199.525) | -484.874( -481.025) | 316.239 | -34.740 | -146.223( -142.374) | -338.651 | |
| 3ns | -257.142( -252.250) | -555.574( -550.682) | 332.083 | -33.651 | -179.019( -174.127) | -376.555 | |
| 4ns | -184.669( -181.096) | -474.791( -471.219) | 323.676 | -33.553 | -144.987( -141.414) | -329.804 | |
| 5ns | -184.836( -180.038) | -480.970( -476.172) | 330.706 | -34.573 | -162.984( -158.186) | -317.986 | |
| 6ns | -215.089( -201.776) | -513.379( -500.066) | 331.582 | -33.292 | -181.463( -168.149) | -331.917 | |
| 7ns | -224.666( -213.083) | -505.049( -493.466) | 314.445 | -34.062 | -170.463( -158.879) | -334.587 | |
| 8ns | -182.786( -178.325) | -453.628( -449.167) | 304.229 | -33.387 | -134.991( -130.530) | -318.636 | |
| 9ns | -231.903( -226.802) | -513.090( -507.988) | 315.202 | -34.015 | -156.808( -151.707) | -356.282 | |
| 10ns | -190.154( -186.687) | -467.055( -463.589) | 310.690 | -33.788 | -140.663( -137.196) | -326.393 | |
| mean | -204.892( -198.527) | -491.332( -484.967) | 320.455 | -34.015 | -158.271( -151.906) | -333.062 | |
| -TdS | 31.779( 29.874) | ||||||
| dG | -173.113( -168.653) kJ/mol = -41.375( -40.309) kcal/mol | ||||||
某蛋白-蛋白: 屏蔽效应显著
| #Frame | Binding( with DH ) | MM ( with DH ) | PB | SA | COU ( with DH ) | VDW | |
|---|---|---|---|---|---|---|---|
| 20ns | -1325.265(-180.626) | -1815.333( -670.694) | 524.813 | -34.745 | -1611.968( -467.330) | -203.365 | |
| 21ns | -1215.585(-109.710) | -1900.597( -794.722) | 723.437 | -38.425 | -1646.821( -540.946) | -253.776 | |
| 22ns | -1452.256(-224.669) | -2026.828( -799.240) | 616.961 | -42.389 | -1740.134( -512.547) | -286.694 | |
| 23ns | -1547.032(-287.347) | -2095.682( -835.997) | 586.848 | -38.199 | -1805.780( -546.095) | -289.901 | |
| 24ns | -1305.430(-188.950) | -1801.379( -684.899) | 535.587 | -39.638 | -1535.285( -418.806) | -266.093 | |
| 25ns | -1407.047(-229.259) | -1940.299( -762.510) | 572.962 | -39.710 | -1670.085( -492.296) | -270.214 | |
| 26ns | -1410.308(-231.828) | -1892.770( -714.290) | 525.772 | -43.310 | -1586.906( -408.426) | -305.864 | |
| 27ns | -1420.565(-214.647) | -2089.784( -883.866) | 715.753 | -46.534 | -1730.488( -524.570) | -359.296 | |
| 28ns | -1429.163(-206.088) | -1972.306( -749.231) | 587.422 | -44.279 | -1679.958( -456.883) | -292.348 | |
| 29ns | -1522.787(-265.283) | -2001.972( -744.468) | 520.315 | -41.130 | -1724.539( -467.035) | -277.433 | |
| 30ns | -1464.768(-260.994) | -1915.157( -711.384) | 494.979 | -44.590 | -1607.751( -403.977) | -307.407 | |
| mean | -1409.110(-218.127) | -1950.191( -759.209) | 582.259 | -41.177 | -1667.247( -476.265) | -282.945 | |
| -TdS | 142.877( 82.579) | ||||||
| dG | -1266.232(-135.548) kJ/mol = -302.637( -32.397) kcal/mol | ||||||
几点思考与想法
- 结合能的计算是一个系统流程, 需要全局考虑, 结合能每项贡献具体计算方法的不同都可能导致总结合能发生变化. 所以在对比评估不同计算流程时, 要考虑到这一点, 不要将不同计算流程的部分贡献拿来组合. 这么做可能会凑出看起来更好的结果, 但也可能只是误差抵消导致的假象. 当然了, 说成杂凑有点低陋, 说成杂化/杂合hybrid就高深多了, 各式论文中屡见不鲜.
- 屏蔽方法对某个特定体系能给出更好结果只是给出了一点线索, 要成为通用的方法可能还不够. 接下来需要对尽可能多的不同体系进行考察, 综合评定其效果. 浙大的侯廷军搜集整理了一个结合能数据库PDBbind, 并在论文[^3]中用于对比评判各种MMGBSA/MMPBSA流程. 同样, 我们可以用这个数据库来测评下屏蔽方法, 如果总体结果确是好于原始的MMPBSA方法, 也算是篇不错的论文. 有了这样的测评结果之后, 可以让人在使用这种方法时更有信心, 当然也可以吸引更多人来使用这种方法.
- 考虑屏蔽效应时, 离子浓度用的是所加盐的浓度(一般为0.15M). 如果模拟过程中没有加盐, 只添加了抗衡离子, 这种情况下还要考虑屏蔽效应么? 此外, 对每一帧NPT轨迹, 严格来说盐浓度也并非固定不变, 我觉得根据每帧轨迹的体积和总离子数目来计算屏蔽长度更说得通.
- 计算MM(COU)时也使用PB方法, 只不过改用不同的介电常数/原子半径, 是否可行? 不知道PB计算时原子半径是否可以设置为零.
- 论文[^4]中提出了一种电荷校正方法, 或许可以看看, 有什么启示.
参考文献
- Samuel Genheden, Ulf Ryde; The MM/PBSA and MM/GBSA methods to estimate ligand-binding affinities; Expert Opinion on Drug Discovery 10(5):449-461, 2015; 10.1517/17460441.2015.1032936
- Hong-ming Ding, Yue-wen Yin, Song-di Ni, Yan-jing Sheng, Yu-qiang Ma; Accurate Evaluation on the Interactions of SARS-CoV-2 with Its Receptor ACE2 and Antibodies CR3022/CB6*; Chinese Phys. Lett. 38(1):018701, 2021; 10.1088/0256-307X/38/1/018701
- Huiyong Sun, Youyong Li, Sheng Tian, Lei Xu, Tingjun Hou; Assessing the performance of MM/PBSA and MM/GBSA methods. 4. Accuracies of MM/PBSA and MM/GBSA methodologies evaluated by various simulation protocols using PDBbind data set; Phys. Chem. Chem. Phys. 16(31):16719-16729, 2014; 10.1039/c4cp01388c
- Martin A. Olsson, Alfonso T. García-sosa, Ulf Ryde; Binding affinities of the farnesoid X receptor in the D3R Grand Challen; J Comput Aided Mol Des 32(1):211-224, 2017; 10.1007/s10822-017-0056-z
