本手册已过时, 不再更新. 如果需要最新手册, 请加入下方QQ群.
分子动力学模拟的一般性步骤
以下是分子动力学模拟的一般性步骤, 具体的步骤和过程依赖于你研究的体系和所用的软件, 但这并不影响我们把它当作一个入门指南.
-
评估体系 首先需要对我们要进行模拟的体系做一个简单的评估, 有三个问题是我们必须要明确的:

-
选择工具 选择合适的模拟工具, 大前提是它能够实现你所感兴趣的目标. 这需要你非常广泛谨慎地查阅文献, 看看别人用这些工具都做了些什么, 有没有和你的研究体系类似的, 相关的研究. 千万不要做到一半才发现原来你用的工具根本就不能实现你所感兴趣的idea, 切记!
在选择合适的模拟工具时, 主要考虑下面两点:
- 软件的选择. 通常与软件主流使用的力场有关, 软件本身也具有一定的偏向性.
- 蛋白体系: GROMACS, AMBER, NAMD均可
- DNA, RNA体系: 首选AMBER
- 界面体系: DL_POLY比较强大
- 材料体系:LAMMPS是不错的选择
- 力场的选择. 力场用来描述体系中最小单元间的相互作用, 是对实验性质或量子化学计算结果拟合后生成的经验式. 有人会嫌它粗糙, 但它确确实实为我们模拟大系统提供了可能, 只能说关注的切入点不同罢了. 常见的有三类力场: 全原子力场, 联合力场, 粗粒化力场. 此外还有所谓第一代, 第二代, 第三代力场的说法, 这里就不一一列举了.
再次提醒注意: 必须选择适合于我们所关注体系和所感兴趣的性质及现象的力场.
- 软件的选择. 通常与软件主流使用的力场有关, 软件本身也具有一定的偏向性.
-
初始结构 通过实验数据或者某些工具得到体系内的每一个分子的初始结构坐标文件. 之后, 我们需要按我们的想法把这些分子按照一定的规则或是随机的放在一起, 从而得到整个体系的初始结构, 这也是我们模拟的输入文件.
-
输入参数 得到了结构输入文件, 我们还需要力场参数输入文件, 也就是针对我们体系的力场文件. 这通常由所选用的力场决定, 包括电荷, 键合参数和非键参数等势能函数的输入参数.
-
确定盒子 体系的大小通常由你所选用的盒子大小决定. 我们必须对可行性与合理性做出评估, 从而确定体系的大小, 这依赖于具体的体系.
-
能量最小化 由于初始构象可能会存在两个原子靠得太近的情况(称之为bad contact), 所以需要在正式模拟开始的第一步对体系进行能量最小化. 比较常用的能量最小化方法有两种, 最速下降法和共轭梯度法. 最速下降法是快速移除体系内应力的好方法, 但是接近能量极小点时收敛比较慢, 而共轭梯度法在能量极小点附近收敛效率高一些. 所以一般做能量最小化时都是先利用最速下降法进行优化, 完成之后再对得到的构象利用共轭梯度法优化一次, 这样做能有效地保证后续模拟的进行.
-
平衡模拟 你需要设置适当的模拟参数, 并且保证这些参数的设置与力场的构造过程相一致. 举个简单的例子, GROMOS力场是用范德华势双截断来定义范德华参数的, 如果你用GROMOS力场的话也应该用双截断来处理范德华相互作用. 常见的模拟思路是, 先在NVT下限制住你的溶质(剂)做限制性模拟, 这是一个升温的过程, 当温度达到你设定的温度后, 接着做NPT模拟, 此过程将调整体系的压强进而使体系密度收敛.
如何判断体系达到平衡是比较技术性的问题. 简单地讲可以通过以下几种方式:
- 看能量(势能, 动能和总能)是否收敛
- 看体系的压强, 密度等等是否收敛
- 看体系的RMSD是否达到你能接受的范围
- 其他经验
-
成品模拟 经过一段时间的平衡模拟, 在确定体系已经完全弛豫之后, 就可以开始采集数据了. 运行足够长时间的模拟以确定我们所感兴趣的现象或是性质能够被观测到, 并且务必确保此现象出现的可重复性.
-
数据分析 数据拿到手后, 很容易通过一些可视化软件得到轨迹动画, 但这并不能拿来发文章. 真正的工作才刚刚开始——分析数据. 你所感兴趣的现象或性质只是表面, 隐含在它们之中的机理才是文章的主题.
GROMACS入门
GROMACS流程图
使用GROMACS对水盒子中的蛋白质进行分子动力学模拟时, 典型的流程图如下.
更详细的例子可参看上面的Getting Started. 运行时可能还需要进行一些能量最小化的步骤: grompp -> mdrun.
| eiwit.pdb |  |
|||||||
| 创建GROMACS的拓扑文件 | gmx pdb2gmx |  |
||||||
 |
 |
|||||||
| conf.gro | topol.top | |||||||
 |
     |
|||||||
| 增大盒子 | gmx editconf |  |
||||||
 |
||||||||
| conf.gro | ||||||||
 |
||||||||
| 填充水, 蛋白质溶剂化 | gmx solvate |  |
||||||
 |
 |
|||||||
| conf.gro | topol.top | |||||||
| grompp.mdp |  |
 |
 |
|||||
| 创建mdrun输入文件 | gmx grompp |  |
||||||
 |
 |
继续运行 | ||||||
| topol.tpr | state.cpt | |||||||
 |
  |
|||||||
| 运行模拟(EM或MD) | gmx mdrun | |||||||
 |
 |
|||||||
| traj.xtc / traj.trr | ener.edr | |||||||
 |
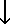 |
|||||||
| 分析 | g_... gmx view |
gmx energy |  |
|||||
另一个适用于GROMACS 4.x的流程图
GROMACS文件类型
GROMACS mdp选项
参见手册第七章运行参数
GROMACS常见问题
GROMACS实例教程
0. 漏斗网蜘蛛毒素肽的溶剂化研究: Amber99SB-ILDN力场(GMX 4.6/5.1)
-
原始文档: John E. Kerrigan 3.3.1版本 4.6版本
参考译文: 梁(leunglm@hotmail.com), 特此致谢.
感谢 陈孙妮 修订翻译舛误之处. - 概述
- 分子动力学模拟
- 模拟结果分析
- 参考文献
- 附录: 更长时间模拟的参数文件
8. 创建周期性体系的拓扑文件: 以石墨烯为例
-
整理: 阮洋; 修订: 李继存
- 概述
- 1. 获取石墨烯的结构文件
- 2. 确定使用的力场
- 3. 准备n2t文件
- 4. 生成拓扑文件
- 5. 检查并修改拓扑文件
- 6. 运行模拟
- 总结
- 参考资料
9. 使用GROMACS计算MM-PBSA结合自由能
10. GROMACS模拟空间非均相体系(板块结构)的并行性能: 区域分解与PME节点设置
-
原始文档: Kathleen Kirchner
翻译: 李继存 - 译者按
- 摘要
- 1. 问题说明
- 2. 研究体系
- 3. GROMACS mdrun文档中的有关说明
- 4. 运行性能测试
- 5. 错误信息
- 6. 如何获得最佳区域分解设置
11. 计算蛋白质不同螺旋之间的夹角
-
整理: 王浩博; 补充: 李继存
- 定义螺旋轴的轴矢量
- 利用
gmx bundle计算不同螺旋之间的夹角 - 参考资料
12. Xmgrace学习笔记
-
整理: 李卫星
- 安装
- 一些资源
- 基本操作
- 附录: 图像文本设置
评论
- 2016-07-14 17:13:42
陈孙妮博主您好~我想问一下,您有中文教程(非中文手册)的PDF版本吗?有的话能发送一份至613674489@qq.com吗~没有的话也万分感谢您的贡献~~ - 2016-07-14 20:23:19
Jerkwin教程没有pdf版, 但你可以直接在chrome中将其保存为pdf, 效果也不差. -
2016-07-21 15:49:42
陈孙妮您好,很抱歉现在才回复,我按您说的做了,可以使用,谢谢您,感恩您所做的一切~ -
2015-06-10 10:29:36
tarot1996博主,我自己再学习过程中也试着翻译了一下教程,打算以后继续翻译更多.可不可以挂到你的网站一起,译的不是很好,你可以先看一下.http://www.lofter.com/blog/tarot1993?act=dashboardclick_20130514_04 -
2015-06-10 12:00:47
Jerkwin热烈欢迎. 你可以把你翻译的教程发给我, 我校对一下就可以放到这里. 如果愿意, 你可以加入我建立的QQ群:132266540. -
2015-06-10 14:06:28
tarot1996群我已经加了,要怎么发给你? - 2015-10-27 15:44:18
kangsgo普通学生可以进群么 - 2015-10-27 17:48:58
Jerkwin谁都可以加入群, 没有什么限制
